上篇文章《欧易打造微生物多样性描述葵花宝典(上)》与大家分享了数据OTU统计、群落结构、Alpha多样性和Beta多样性方面的描述建议,本篇继续打造差异筛选、关联分析和功能预测的超强攻略,相较于上篇基础多样性等方面的分析内容,下篇主要围绕差异及功能等方面展开,也是文章撰写的核心目标。
一、差异筛选
通过筛选与不同比较组差异显著菌群,获得与研究相关的重要标志物。本部分结果是微生物多样性的核心,其中筛选到的微生物与研究目的直接相关,其功能和机制也有待进一步挖掘。多组组间差异显著性计算常用ANOVA、Kruskal Wallis算法,当数据不满足正态分布,ANOVA分析则相对来说并非太合适,Kruskal Wallis检验就能针对不满足正态分布数据,进行组间比较。两组组间差异显著性计算常用Wilcoxon、T-Test算法。筛选标准默认为p<0.05,也可以根据实际情况继续缩小范围,采用更小的p值或FDR来过滤。常用的差异信息采用箱线图或Heatmap图来呈现(如下图),都是基于相对丰度绘制,选其一即可,常见的差异物种分类水平为属水平。
描述方法可以考虑描述具体算法和筛选方式,针对关注物种进行深入讨论,例如在Kruskal Wallis算法中,根据FDR<0.05筛选到大肠杆菌有显著性变化,通过文献和数据库查找,确认其有哪些功能,后续通过验证手段进一步探索机制。
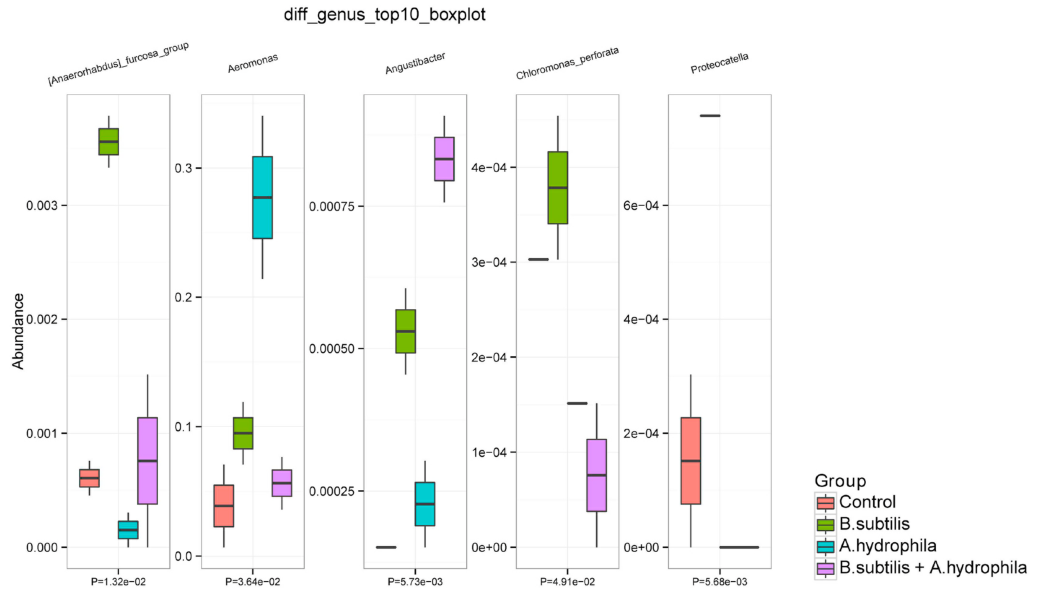
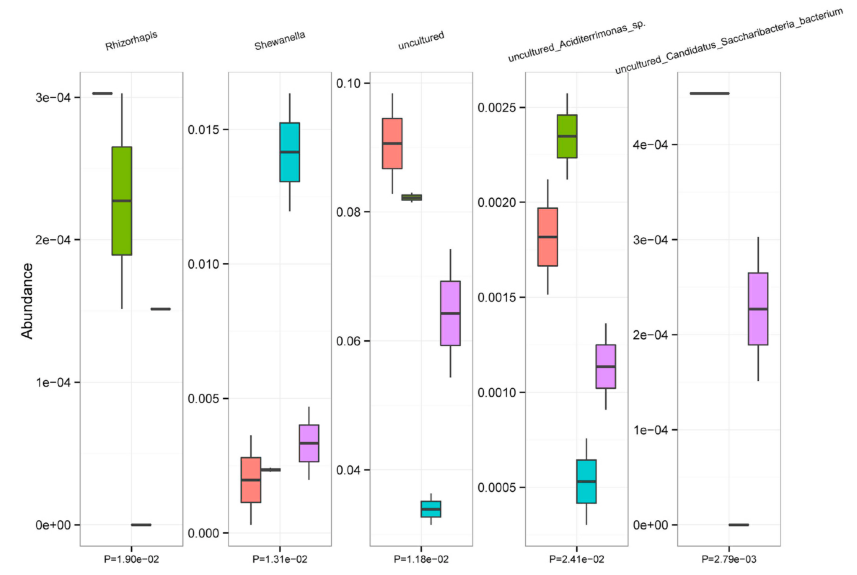
(上图描述示例:图中显示有显著差异的前10个属的相对丰度(Kruskal-Wallis,p<0.05)。结果发现,Aeromonas和Shewanella的相对丰度均高于对照组[1]。)
LEfSe分析在文章中也较常用(如下图),用于揭示两组或以上生物群落差异物种的组成,结果展示包括三部分,分别为差异物种对差异贡献度大小分析、差异物种注释分析和差异物种在每个样本中的相对丰度情况,适用于后续biomarker研究的筛选。贡献度大小根据LDA Score来评判,大于默认阈值的分类单元,即不同组中丰度差异显著的分类单元,默认阈值2.0,也可以根据具体情况调整,值越大越显著。
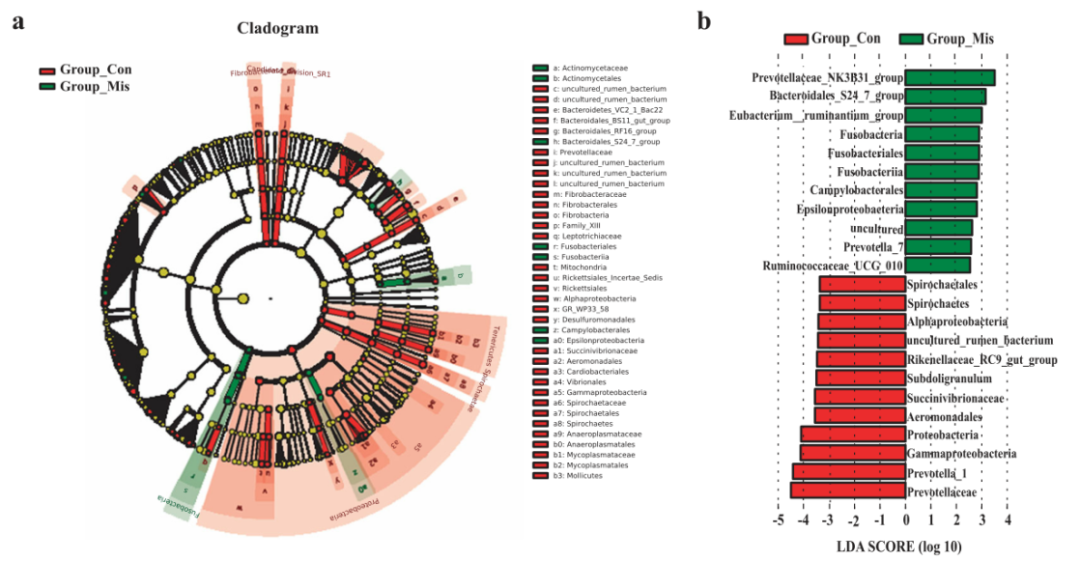
(上图描述示例:Group-Mis中的几种条件致病菌(包括Prevotellaceae_NK3B31_group, Bacteroidales_S24_7_group 和 Eubacterium ruminantium_group)均显著偏高(LDA评分(log10) >3.0),而Prevotellaceae, Prevotella_1 和 Gammaproteobacteria是对照组中最丰富的微生物群(LDA评分(log10) >4.0)。这些结果表明,粪便微生物群组成的变化与流产有关[2]。)
二、关联分析
在采用不同的分组比较差异微生物后,如何与生理指标进行关联,是很多研究的重中之重,关联分析就能很好的解决这个问题。通过将微生物数据与其他信息计算相关性等方法,探讨微生物对生理生化影响及组学互作等情况。可关联的数据包括:物种之间、物种与表型数据、生理指标、代谢物丰度等,计算不同指标与微生物丰度之间的相关性和显著性,相关性系数越高越好,显著性p值越小越好。
文章中常见的呈现形式如相关性Heatmap图、网络图和CCA/RDA分析。Heatmap图(如下图)主要针对物种样本间的相对丰度与表型等进行Spearman相关系数计算得出,根据结果可获得表型与目标微生物的正负相关性,进而引入后续更深入的功能研究。
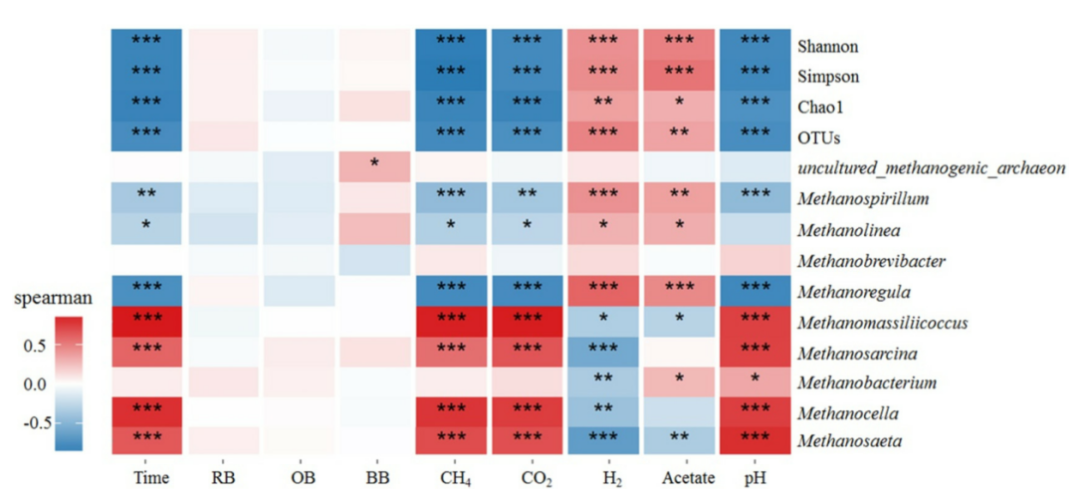
[3]
RDA/CCA 分析可以检测环境因子、样品、菌群三者之间的关系或者两两之间的关系(如下图)。
描述方法可以根据微生物与环境因子等信息的相关程度来展开,例如某优势微生物与哪些指标正相关,哪些指标负相关,差异变量的关联物种有哪些,具体的筛选显著性p值或FDR是多少。
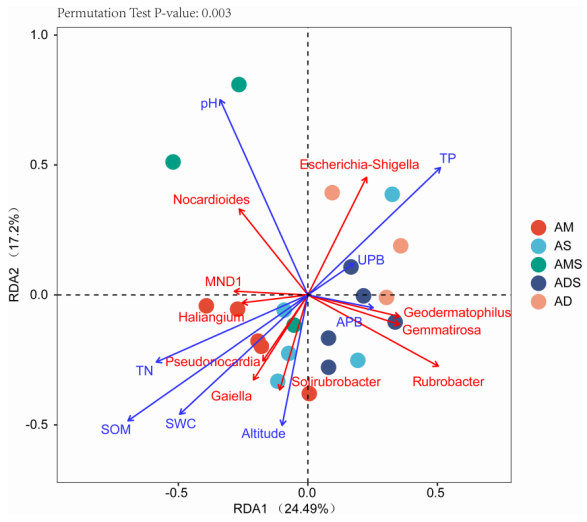
(上图描述示例:该图是描述细菌、理化指标和样本间的关系,揭示了环境变量对土壤细菌群落的解释率为41.69%,第一轴和第二轴的贡献率分别为24.49%和17.2%。分析结果还揭示土壤细菌各属与环境变化的相关性,不同土壤属的主导因子不同。例如,土壤pH与Nocardioides, Escherichia-Shigella, MND1 和 Haliangium呈正相关,与 Rubrobacter, Solirubrobacter, Gemmatirosa 和 Geodermatophilus呈负相关。此外,采样点的差异也可以通过环境变量反映出来,不同采样点的土壤细菌丰度也表现出差异性。例如,AM中的相应样本在Pseudonocardia向量上的投影点在AD的投影点之前,表明AM中的Pseudonocardia比AD具有更大的丰度潜力。由此可见环境因子,特别是SOM、TN、TP、pH和SWC与土壤细菌群落关系密切(p<0.05)[4]。)
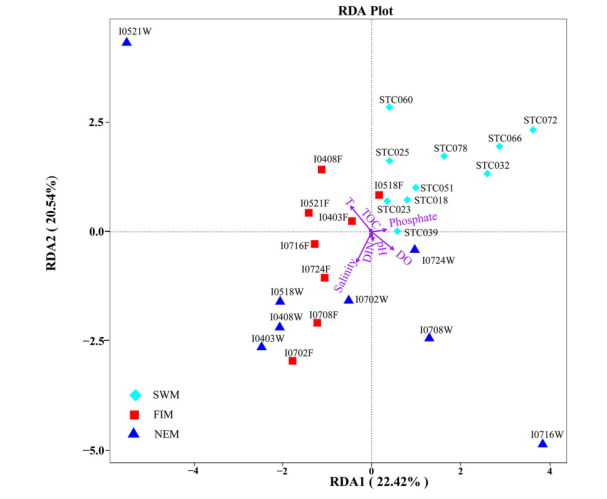
(上图描述示例:该图是描述理化指标和样本间的关系,通过RDA证实,第一轴和第二轴解释物种水平表面细菌群落和环境因子累积方差的42.96%。温度、盐度、溶解氧和其他参数的p值分别为0.0015、0.0005、0.006和>0.32,由此说明温度、盐度和溶解氧是影响细菌群落的主要因素[5]。)
三、 功能预测
功能预测是通过预测已知微生物基因功能的构成,从而统计不同样本和分组之间在功能上的差异。细菌常用PICRUSt功能预测分析,真菌常用FunGuild功能预测分析。该部分分析仅为预测结果,因此会有被编辑质疑的可能,建议附加宏基因组测序、荧光定量等手段,验证功能基因以进一步证实结论。实际分析和实验结果反映该预测的准确性和样本有很大关系,其中肠道菌群和土壤菌群这些研究比较多的菌群,其预测准确度会相对高一些。常用的功能数据库是KEGG数据库,结果主要以Heatmap图、柱形图的形式展示(如下图),值得注意的是,预测结果不能精确到某一物种,只能进行通路水平的预测和丰度分析。
描述内容主要根据关注功能展开,例如在不同比较组中某几个通路有显著差异,推测在不同处理其功能相关微生物群是有区别的。
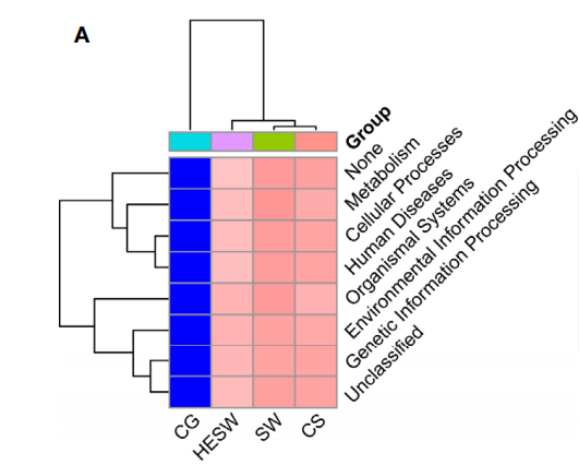
(上图描述示例:根据PICRUSt发现与CG组相比,来自其他组属于BK患者眼睛微生物具有显著不同的细菌基因
功能组成。BK患者的眼部微生物组呈现出明显更多与代谢、细胞过程、人类疾病、生物系统、环境信息处理和遗
传信息处理相关的基因,也就是说BK患者的眼部微生物群显得更加活跃和具有攻击性[6]。)
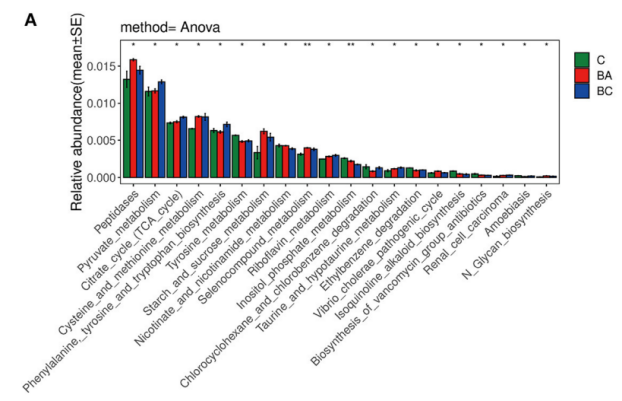
(上图描述示例:通过PICRUST和KEGG富集研究,不同丰度的微生物主要富集在肽酶、丙酮酸代谢和TCA循环等代谢途径中[7]。)
以上三部分涉及到分析小工具链接如下,老师可以根据需求进行个性化处理:
LefSe分析小工具链接:https://cloud.oebiotech.cn/task/detail/LEfSe-oehw/
差异筛选小工具链接:https://cloud.oebiotech.cn/task/detail/diffstatistic-oehw/
相关性分析小工具链接:https://cloud.oebiotech.cn/task/detail/correlation-multiomics-oehw/
关联散点图小工具链接:https://cloud.oebiotech.cn/task/detail/scatterplot-multiomics-oehw/
PICRUSt2功能预测小工具链接:https://cloud.oebiotech.cn/task/detail/PICRUSt2-oehw/
总结上下篇共七部分内容,虽然文章不要求全部描述,但每部分都有各自特点和意义,老师可根据研究目的规划组合方案。针对筛选到的差异微生物,可以根据文献、数据库等信息获得其研究进展,进一步探索其作用机制和前景。如想提高文章水平,可以考虑结合宏基因组、菌群移植、多组学联合等手段,系统阐述作用机制和功能。其他个性化展示形式或高级分析需要根据具体需求选择,此处不做深入展开。希望能给各位老师文章撰写过程提供帮助。
注:本文内容仅供示例参考,其中涉及的结果内容描述和分析方法会有更多呈现形式,建议老师根据研究目的和结果进行最终判断,如有不妥之处欢迎随时交流。
参考文献
近期活动
活动一:微生物多样性测序优惠大放送

活动二:宏基因组产品钜惠来袭







