前不久,咱们给老师们介绍过基因表达可视化的多种实现方式,有windows操作的IGV,也有Linux环境下使用的工具。很快小欧在后台收到很多老师的需求,希望有更详细IGV教程。于是小欧马不停蹄赶工出来,咱们一起来看看吧~
IGV介绍
1、简介:Integrative Genomics Viewer (IGV) 是一种高性能、易于使用的交互式工具,用于对基因组数据进行可视化探索。它支持所有常见类型的基因组数据和metadata的灵活集成。
2、功能:IGV支持各种各式的大型数据集的可视化,包括二代测序,芯片测序数据和基因组数据等。例如,查看转录组数据不同区域的Reads丰度,不同样本的基因表达差异,查找SNP位点,查看甲基化,查看基因结构信息。
IGV 软件安装
安装包下载地址:
https://software.broadinstitute.org/software/igv/download
官网下载即可,推荐使用内置java版本,以方便安装,如下图:
IGV 有下载文件的需求,所以要通过网络访问权限哦,然后根据提示一步步安装即可。
下载好的IGV主窗口如下:
IGV软件界面介绍
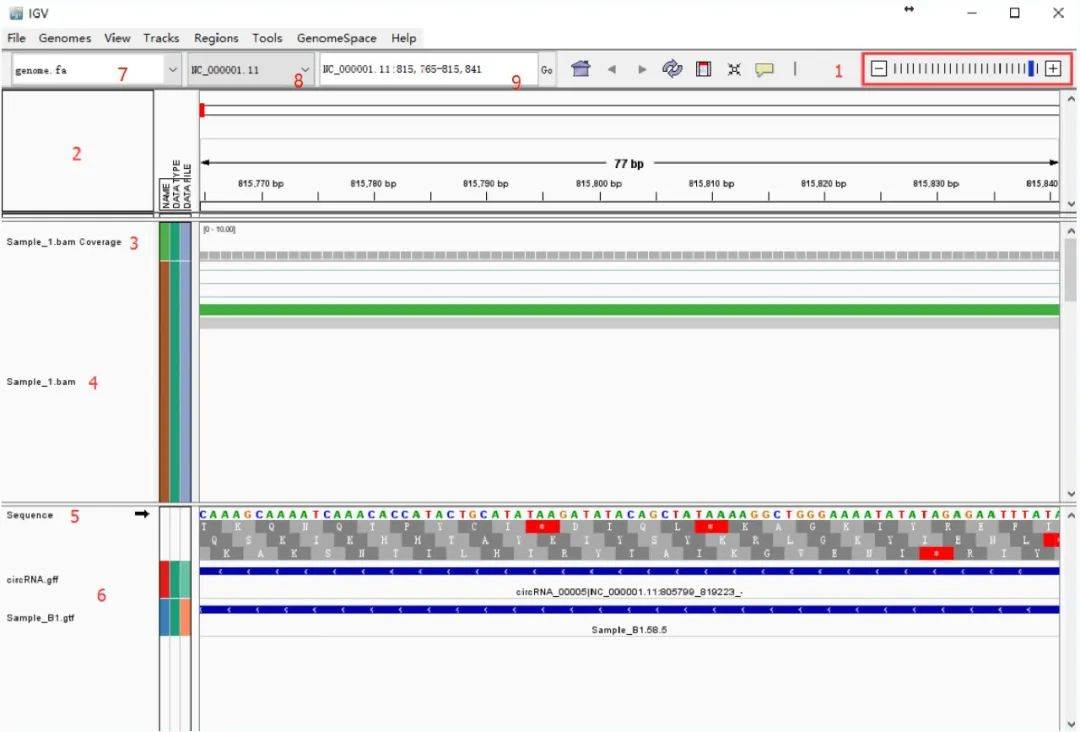
1、设置放大倍数,设置在5-8之间可以比较清晰的查看reads的分布状况,也可以通过双击选中区域进行放大;
2、Fa 文件栏,显示物种的Fasta文件,也就是基因组文件;
3、Read coverage: reads的覆盖度,准确表现该次测序reads的覆盖度;
4、样品bam文件:本次测序的reads经与参考基因组比对后的结果,可得到其在染色体上的分布情况;
5、Sequence:注释文件的核酸序列信息;
6、GTF注释文件的注释信息,此列展示注释文件中的基因及外显子的信息
7、选择参考基因组,IGV上已有一部分物种基因组。
8、可以选择基因组染色体,是一条还是全部。
9、显示特定位点或特定基因,可对关注的基因及基因集进行直接搜索。
查看转录组的比对数据
1、导入基因组
既可以导入IGV自带的参考基因组文件,也可以从本地导入已下载好的参考基因组文件。
导入IGV自带的基因示例:
可在基因组选择框中选择自己数据对应的基因组,或下拉框中输入关键词选择对应的基因
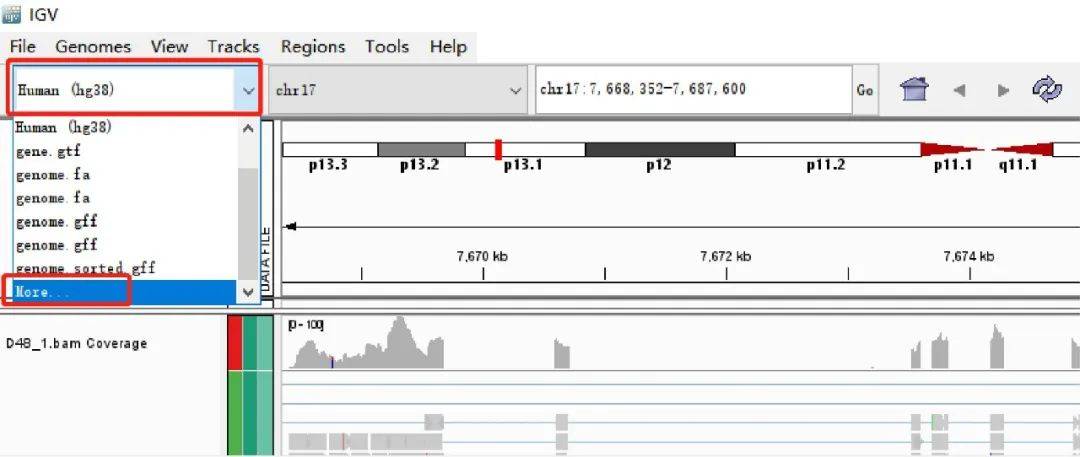
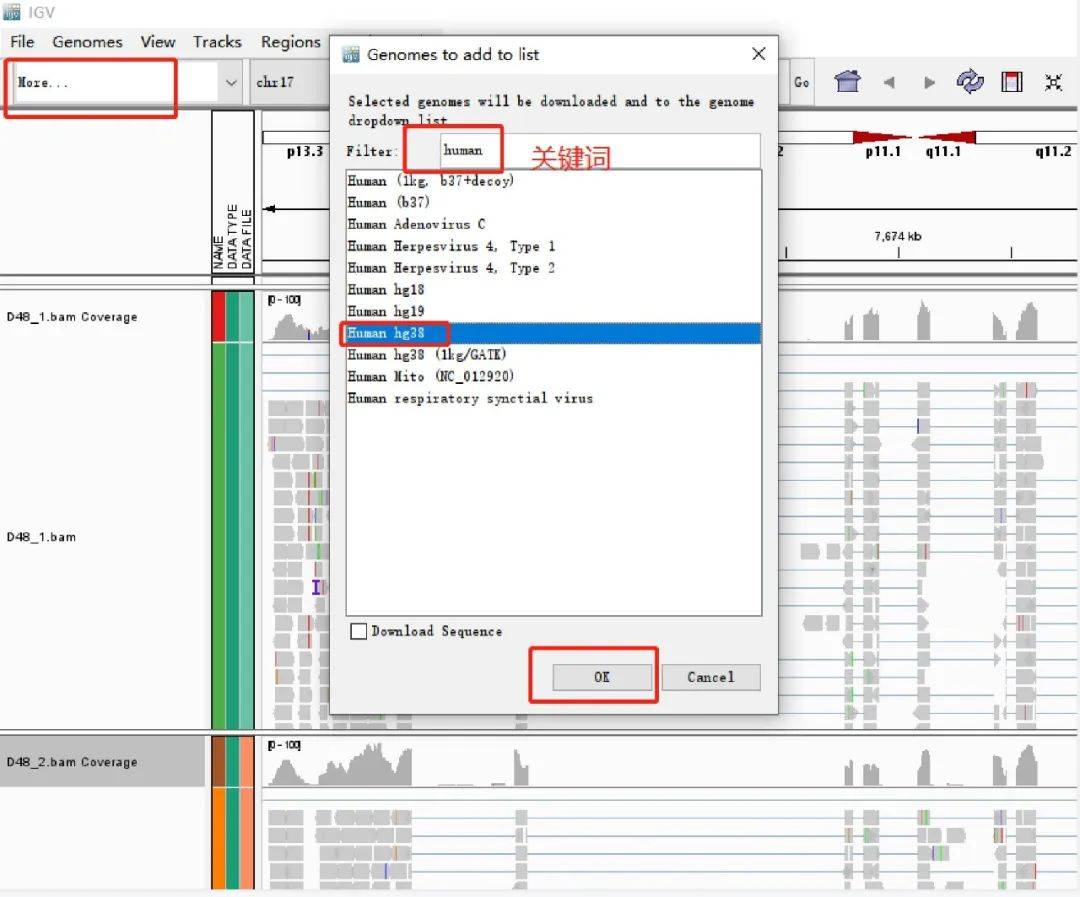
从本地导入已下载好的参考基因组示例:
从本地导入基因组文件的同一路径,还要准备好基因组的gtf注释文件或者gff注释文件,基因组文件通过Genomes→Load Genome from File... 加载,注释文件需要通过File→Load from File... 加载;加载注释文件过程中会有提示需要建立gff文件的索引,可根据提示操作即可。
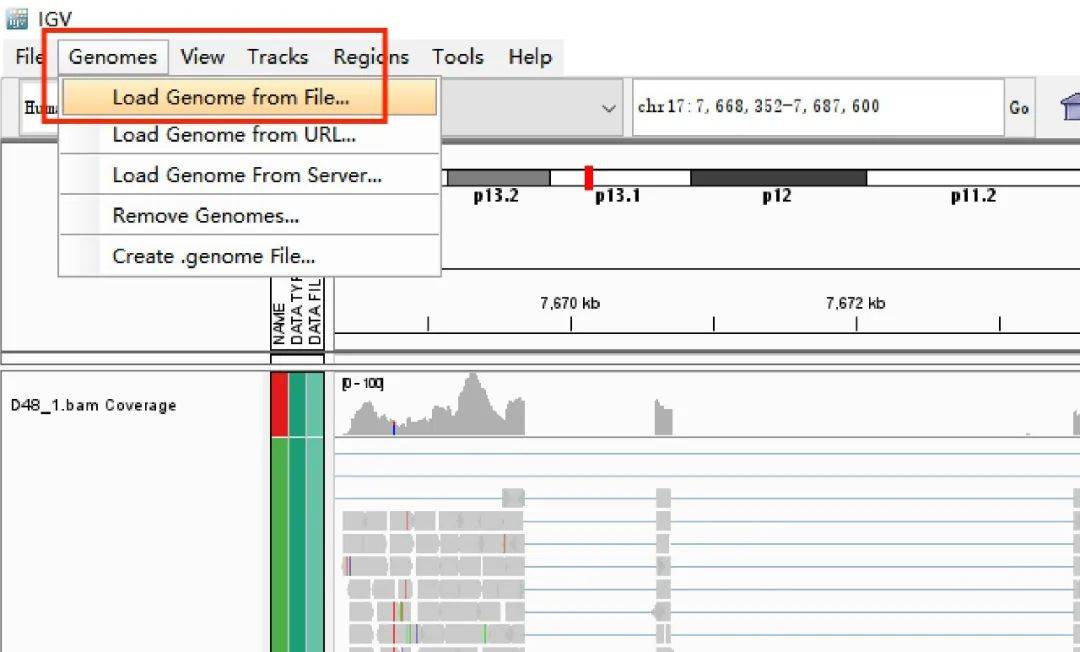
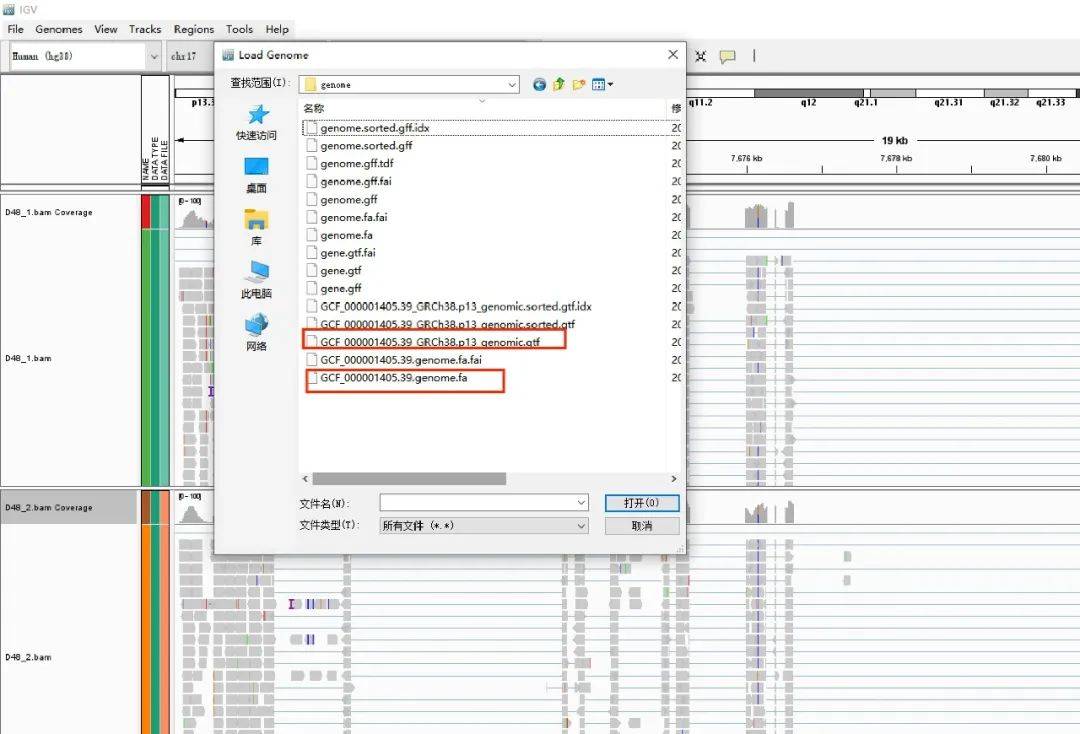
2、导入bam 文件
bam文件也是通过File→Load from File... 加载,准备bam文件时,要在相同目录下同时存在bam.bai文件。
3、查看样本的表达情况,可以通过不断放大关注位点查看更多详细信息。
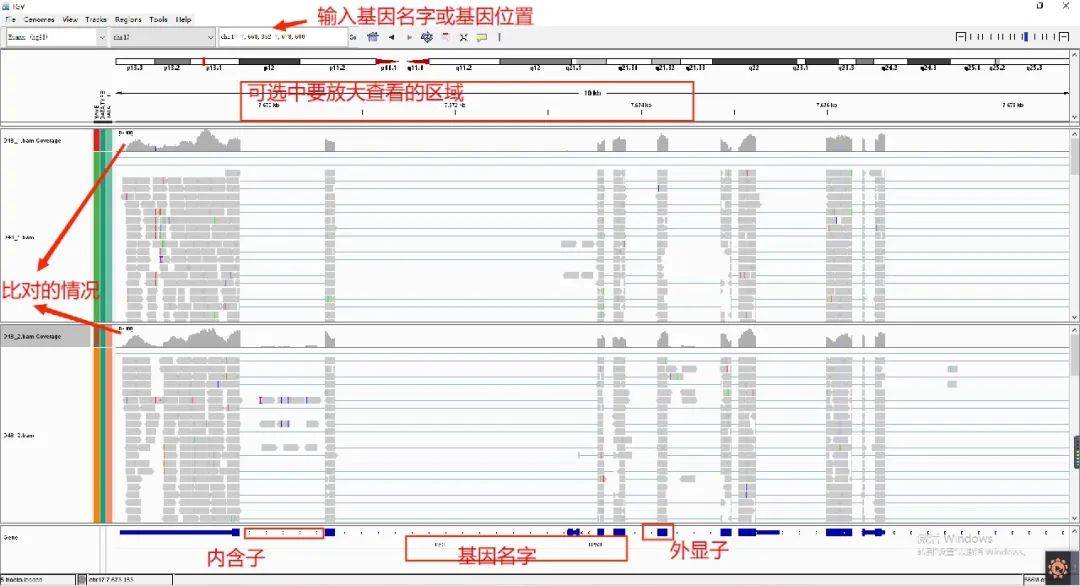
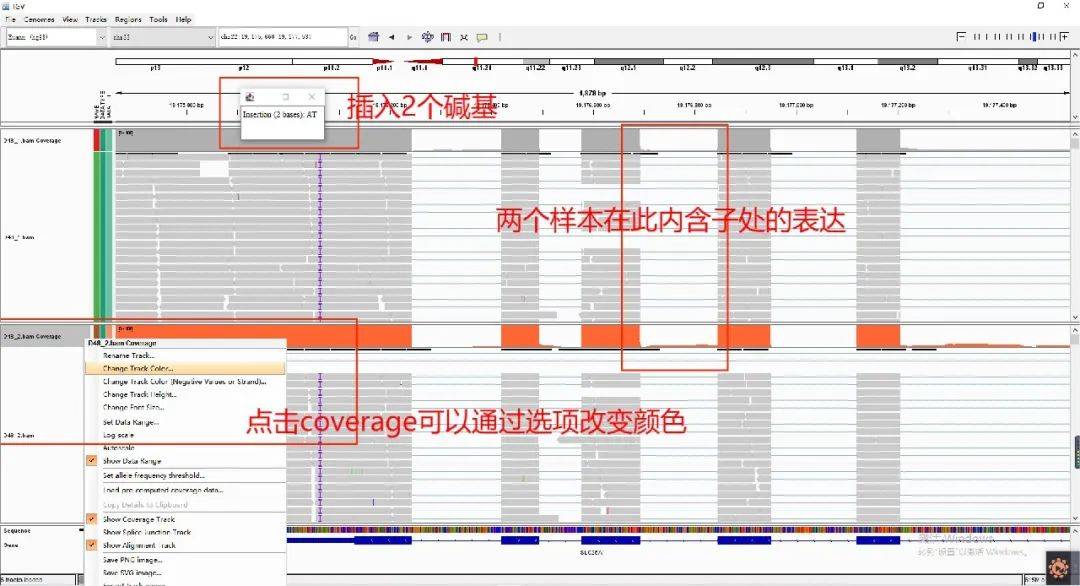
注意:
要选择自己对应的参考基因组文件,且基因组文件较大,加载需要一定的时间。
选择的基因组区域过大,reads覆盖区会出现“Zoom in to see coverage” 的字样,只需增加放大倍数即可。
更多教程
IGV的用途很多,在这里不再做详细的介绍,更多功能可参考:
1、欧易生物转录组分析报告中的
result/3.supplemental_material/IGV
使用说明.html
2、官网用法介绍:
http://software.broadinstitute.org/software/igv/UserGuide
3、详细视频介绍:
https://pan.baidu.com/s/1Cwnlr5Our9lTRGViJCrWcQ
提取码:g6h3

长按扫码
添加客服询问相关问题
猜你想看
3、“码”辅导——微生物样本有其他来源的污染怎么办?Decontam开源包帮你忙!
4、叮叮叮~新知识已到达!教你用scMetabolism做单细胞代谢分析
End本文系欧易生物原创






