精子发育是所有有性生殖物种中最复杂的过程之一,需要复杂的转录后基因调控(post-transcriptional gene regulatory,PTGR),以确保产生功能完善的雄性精子。RNA结合蛋白(RNA-binding proteins,RBP)是PTGR机制的组成部分,通过协调RNA活动的各个方面,包括运输、定位、稳定性等以维持其稳态。DEAD-box(DDX)蛋白家族是已知最大的RNA解旋酶家族,已知参与调节RNA代谢的各个方面。在生殖系统中,DDX20对小鼠卵巢形态和功能的调节至关重要。然而,它在男性生殖系统中的作用仍处于未知状态。本研究旨在探讨DDX20在男性生殖系统发育中的分子机制。
2024年4月,中国医学科学院基础医学研究所宋伟团队、余佳团队联合美国加州大学闫威团队在《Developmental Cells》(IF:11.8)上发表了题为“DDX20 is required for cell-cycle reentry of prospermatogonia and establishment of spermatogonial stem cell pool during testicular development in mice”的研究论文。该研究通过基因敲除、免疫组化、微量Ribo-Seq、RNA-Seq和微量蛋白质组学揭示DDX20通过与核糖体蛋白质相互作用,调节细胞周期相关蛋白的表达,促进T1-ProSG(有丝分裂静止的前精原细胞)重新进入细胞周期和恢复增殖,进而促进精原干细胞(spermatogonial stem cell,SSC)池的形成。

发表期刊:Developmental Cells
影响因子:11.8
材料:DDX20条敲鼠、野生型小鼠、小鼠精原干细胞、HEK293T细胞
主要技术方法:免疫组化,免疫荧光,qPCR,WB,Tunel,co-ip,PLA(邻近相互作用分析),蛋白质合成检测,活细胞单分子成像、基因敲除、RIP-Seq,SMART-Seq,微量Ribo-Seq、微量蛋白组
其中微量蛋白质组学由欧易生物质谱平台提供技术支持
技术路线
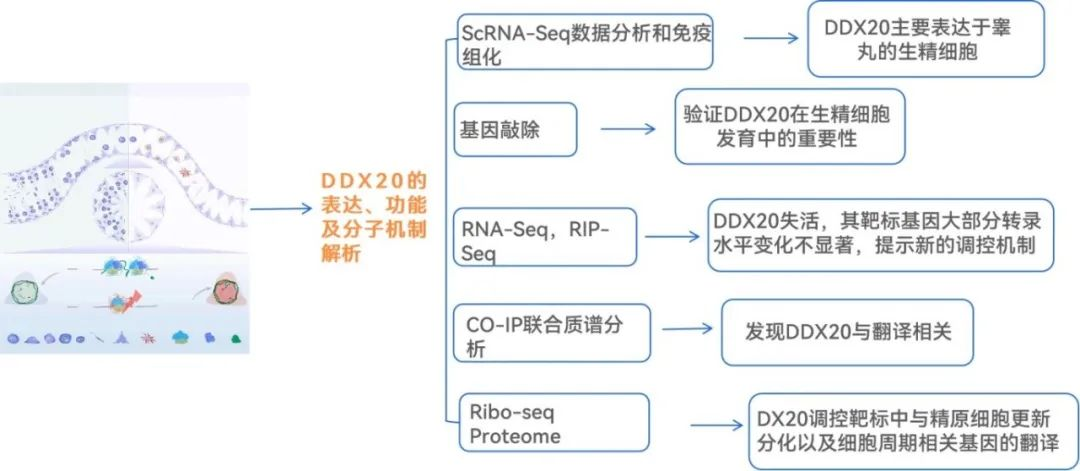
研究结果
1. RBP在精子发育中具有潜在功能
为确定在精子发生中关键作用的RBP,作者从文献中收集了4692个人类RBP。然后,针对人类蛋白质组图谱的转录组数据集,进行了加权共表达网络分析。共鉴定出19个模块。模块5包含514个RBP,显示出最高的睾丸特异性。进一步系统分析,共发现了30个未被报道的具有潜在生精功能的RBP,其中9个与不育相关。ScRNA-seq数据分析发现,无精子症患者睾丸中DDX20、ODF2等基因的表达水平显著降低。另外,DDX20被报道为非阻塞性无精子症的生物标志物,说明DDX20在睾丸生精过程中发挥着重要作用。
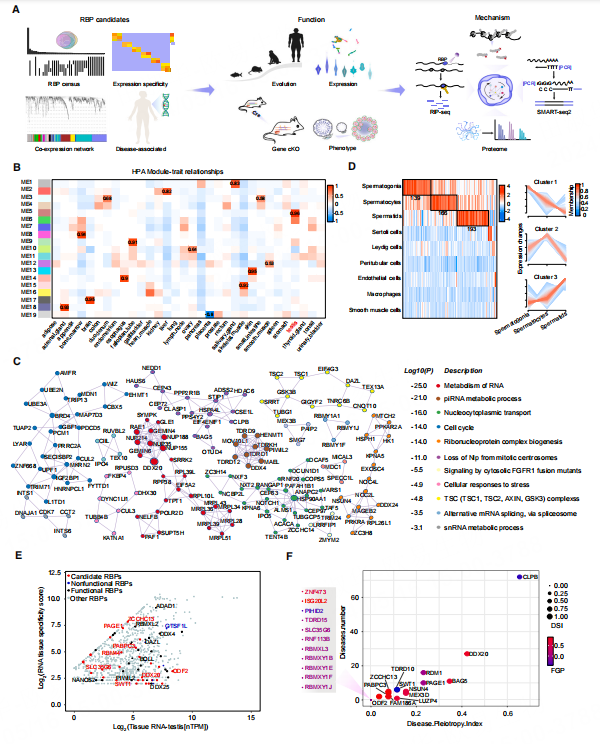
图1 许多RBP优先在人类睾丸中表达,在精子发生中具有潜在的功能
2. DDX20主要表达于睾丸的生精细胞中
DDX20是SMN复合物的一个组成部分,该复合物已被证明参与了小鼠精原细胞的维持。通过分析多组睾丸scRNA-seq数据集,研究者发现DDX20主要表达于生精细胞而不是睾丸体细胞,qPCR和IHC证实了这一结果。免疫荧光成像结合细胞亚组分分离后检测分析进一步验证了DDX20在小鼠睾丸中的分布位置,即精原细胞,精母细胞的细胞质和圆形精子细胞中的细胞核。
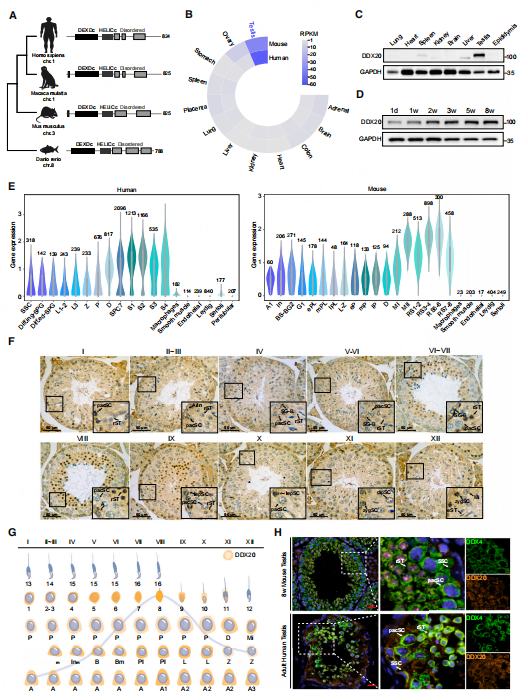
图2 DDX20是一种进化上保守的蛋白,主要在睾丸的生精细胞中表达
3. DDX20 对于精原细胞的激活和精子发生的周期是必需的
随后作者使用DDX20基因敲除鼠验证DDX20在精子生成中的重要性。研究发现,敲除DDX20的小鼠在出生4天后生精细胞开始减少,7天后大部分生精管腔中无生殖细胞,出生21天后完全丢失生殖细胞,表现为临床唯支持细胞综合征表型。进一步分析发现DDX20 条件性敲除小鼠残留的生殖细胞均为未分化的MKI67阴性的精原细胞。而且,代表精原干细胞池形成的胞核表达的FOXO1依然在胞质表达。这些结果表明DDX20 缺失后,导致前精原细胞(ProSG)在小鼠出生后无法恢复增殖形成精原干细胞池。
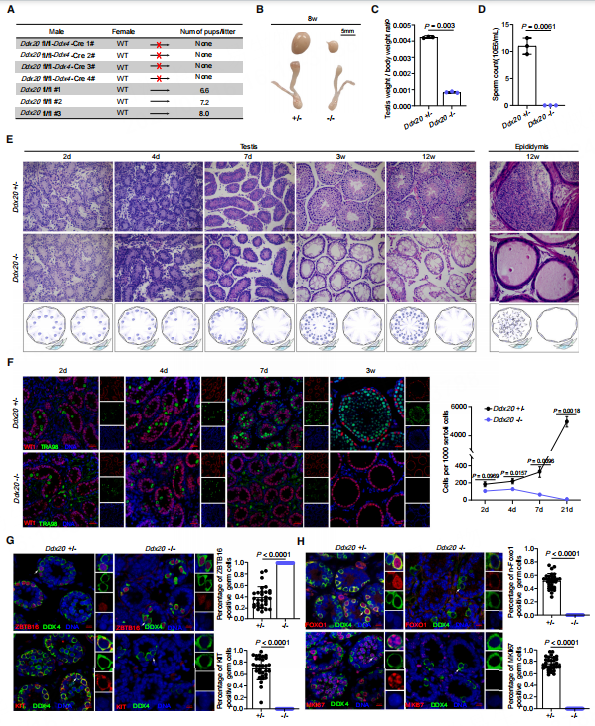
图3 DDX20是小鼠早发原细胞激活和精子发生的第一个周期所必需的
4. DDX20失活导致精原细胞转录组变化
接下来作者对睾丸中纯化的精原细胞进行SMART-seq测序,发现参与精原细胞稳态、DNA复制和细胞周期的40个代表性基因均下调,这一发现与在DDX20缺失的T1-ProSG中观察到的G0/G1细胞周期阻滞相一致。T2-ProSG标记物ETV5表达水平显著低于P2对照睾丸。这些数据进一步支持了在DDX20缺失小鼠中观察到的主要表型,即T1- ProSG未能重新进入细胞周期,并在尝试分化为SSCs和/或祖精原细胞失败后被耗尽。
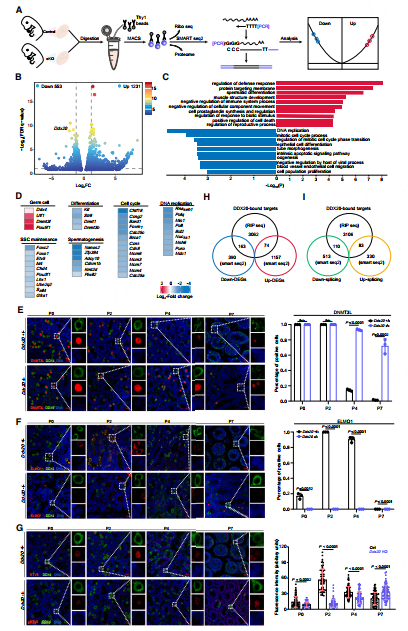
图4 DDX20失活导致精原细胞转录组学变化
为了鉴定DDX20的靶点,使用P7(出生7天) WT小鼠和DDX20 KO小鼠睾丸进行了RNA免疫沉淀,然后进行了高通量测序。发现只有7%的DDX20靶标在DDX20缺失时发生失调,表明DDX20对其靶转录本的稳定性影响很小或没有显著影响。为了确定DDX20失活是否对精原细胞中其靶mRNA的选择性剪接有影响,使用RNA-seq数据集进行了转录本剪接复制多元分析(replicate multivariate analyses of transcript splicing,rMATS),以确定选择性剪接事件(ASEs)。研究发现,大多数对DDX20消融反应的失调基因没有参与ASEs,只有一小部分的DDX20靶mRNA与ASEs相关。这些数据表明DDX20作为RBP可能在细胞质中具有未知的功能。
5. DDX20通过与SMN相互作用,促进核糖体翻译
作者通过CO-IP联合质谱分析来研究与DDX20相互作用的蛋白质,发现除了SMN复合体的成员外,DDX20相互作用组存在大量核糖体和翻译相关蛋白的富集。另外,通过在小鼠睾丸组织细胞及HEK293T细胞中对这些相互作用的蛋白进行验证,发现DDX20确实与核糖体/翻译相关蛋白相互作用,包括RPS8(核糖体蛋白)、RPL4(核糖体蛋白)等。鉴于SMN与主动翻译核糖体相关,接下来研究DDX20和核糖体之间的相互作用是否依赖于SMN。为此,敲除HEK293T细胞中的DDX20或SMN,以检测大核糖体亚基RPL4与DDX20或SMN的结合能力。结果显示,在不影响SMN蛋白水平的状态下,DDX20抑制削弱了SMN-RPL4的相互作用。而SMN的下调影响了DDX20的蛋白水平,但对DDX20与RPL4的结合能力影响有限,表明DDX20对SMN与核糖体的结合至关重要。
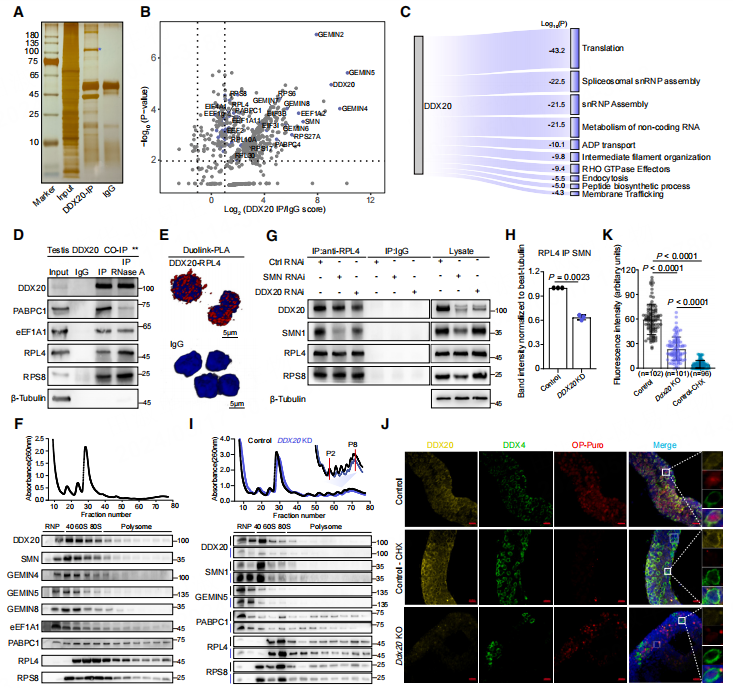
图5 DDX20是一种核糖体相互作用蛋白,调节蛋白质合成
为了进一步验证DDX20在蛋白质合成中的作用,在P4时期从DDX20 KO小鼠和对照小鼠中分离曲细精管,进行OPP-Click-iT反应,检测蛋白质的合成。免疫荧光图像显示,在DDX20 KO小鼠的DDX4阳性生殖细胞中,OPP掺入受到抑制,反映了蛋白合成率的下降。综合这些结果,强烈暗示DDX20作为一个真正的与核糖体相关的蛋白,与SMN和核糖体相互作用,调节翻译过程。
6. DDX20的消融破坏了精原细胞中一个细胞周期相关基因子集的翻译
为验证DDX20在精原细胞中mRNA翻译的作用,对精原细胞进行了微量Ribo-seq测序。发现DDX20-KO精原细胞中正在翻译的RNA片段(RPFs)有5114个上调和2949个下调。另外,KO组中RPFs在前28个密码子上富集,表明DDX20的敲除导致核糖体占用减少,最终导致翻译下调。将DDX20-KO精原细胞的翻译组和转录组进行重叠,发现904个翻译效率(TE)下调的基因和226个TE上调的基因,其中260个下调基因和63个上调基因为DDX20的靶基因。GO富集分析显示,上调基因主要参与GTPase周期、赖氨酸降解等,而下调基因主要参与有丝分裂细胞周期、染色质组装、干细胞维持等。
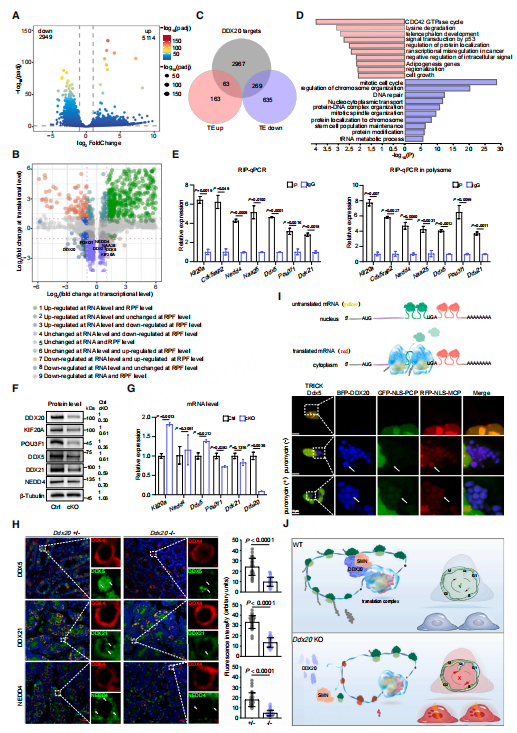
图6 DDX20缺失损害了精原细胞中一个基因子集的翻译
对上述同一批实验进行微量蛋白质组学分析,发现有883个上调蛋白和4265个下调蛋白。进一步的翻译组和蛋白质组的比较分析发现,一些在翻译水平上上调的基因在蛋白水平上被下调。然而,第七模块的基因(RPF上调,蛋白质下调)在KO组显示出不同位置的核糖体暂停,表明DDX20不仅导致部分基因的核糖体占用减少,还导致翻译暂停。此外,还发现蛋白质组中上调的基因主要富集于生物过程,包括细胞应激、蛋白定位、蛋白质折叠等,这表明DDX20缺乏导致翻译下调,最终导致蛋白质稳态失衡。
研究结论
本文通过系统性分析筛选获得与不育相关的生精功能性RNA结合蛋白质-DDX20,分析ScRNA-Seq数据集结合多种分子实验发现了DDX20主要表达于睾丸的生精细胞中,通过基因敲除揭示了其在精原干细胞发育形成中的重要功能,通过SMART-Seq联合RIP-seq发现DDX20可能存在潜在未被发现的分子调控功能,通过CO-IP联合质谱分析,发现了DDX20作为新的翻译调控因子在蛋白质合成中具有重要作用,最后结合微量Ribo-seq联合微量蛋白组及活细胞单分子翻译成像技术发现DDX20促进其靶标基因中与精原细胞更新维持以及细胞周期相关基因的翻译。
本研究工作得到中国医学科学院医学与健康科技创新工程(2021-I2M-1-019、2021-I2M-1-066)、国家重点研发计划(2022YFA0806302)和国家自然科学基金(92268111、32370910、31970794和32000586)等项目的资助。基础医学研究所宋伟研究员、余佳研究员和美国加州大学闫威教授为该论文共同通讯作者。基础所邹定峰博士后、李凯副研究员和博士研究生苏路瑛为该论文的共同第一作者。
原文链接:
https://pubmed.ncbi.nlm.nih.gov/38657611/






