本篇为山西医科大学第一医院以Xiao Yu,Xinri Zhang为通讯作者在Microbial biotechnology杂志发表题为“Unravelling the enigma of the human microbiome: Evolution and selection of sequencing technologies”的研究论文,该研究总结了扩增子测序、宏基因组测序、靶向测序以及两项微生物研究领域新技术2bRAD-M简化微生物组技术和MobiMicrobe技术的优缺点。

研究背景
扩增子测序(Amplicon Sequencing)、宏基因组测序(Metagenomic Next-Generation Sequencing, mNGS)和靶向测序(Targeted Next-Generation Sequencing, tNGS)是微生物组研究中最主流的DNA测序技术。但是扩增子测序的分辨率较低,一般只能达到属水平,而mNGS需要较高DNA起始量,且容易受到宿主DNA的干扰。虽然tNGS可以排除宿主核酸干扰,但不能识别数据库中没有的新病原微生物。最近新出现的两项技术MobiMicrobe和2bRAD-M简化微生物组技术有助于填补这些空白,为更精确的菌株水平微生物基因组研究成为可能。鉴于测序技术和策略的快速发展,对这些方法的总结和分析至关重要,可以让研究人员了解不同技术的优缺点,从而选择最适合他们研究的方法。
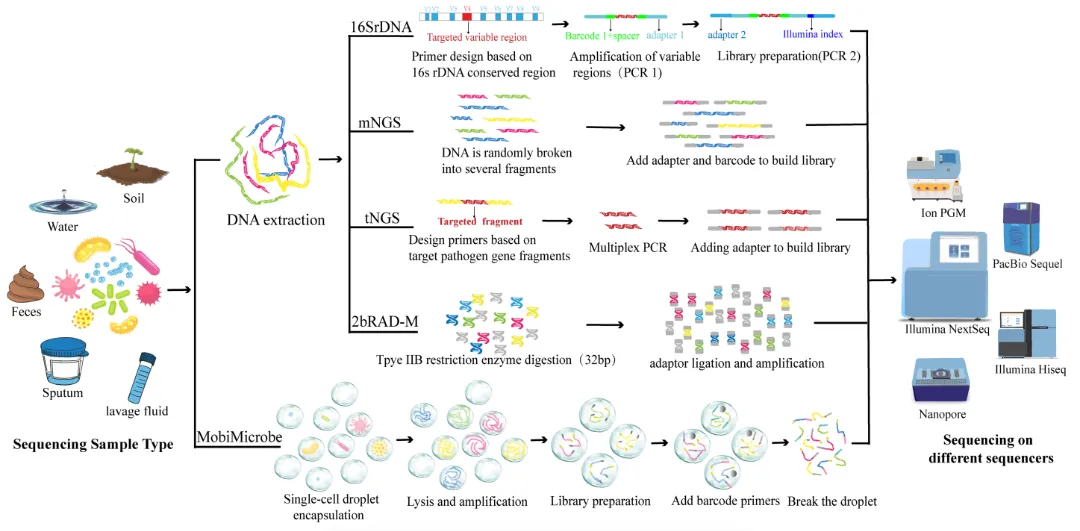
图1 五种微生物测序技术的流程示意图
分为三个阶段:样品采集,样品处理和文库构建以及高通量测序
01、扩增子测序:进化、应用和未来
扩增子测序主要用于微生物群落的物种组成、物种间的进化关系以及微生物群落的多样性研究。扩增子测序中使用的主要标记基因包括原核生物的16s rDNA(图2A)、真核生物的18s rDNA(图2B)和内部转录间隔子(ITS)。扩增子测序经历了以Sanger测序为代表的第一代测序技术、以SBS(Sequencing by Synthesis)测序为代表的第二代测序技术(SGS)和以单分子纳米孔DNA测序或单分子实时测序(SMRT)为代表的第三代测序技术(TGS),扩增子测序几乎可以应用于任何样品。
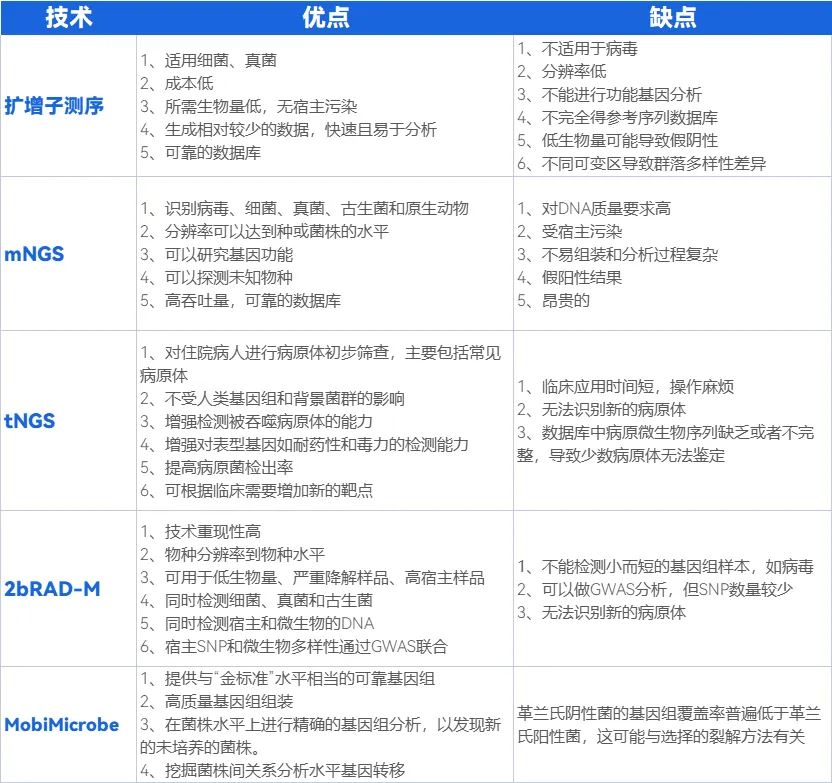
扩增子测序是一种无需培养即可检测微生物的精确方法。已成为诊断微生物学的重要参考,并解决了细菌性病原体鉴定中的许多问题。该技术成本低、分析快、有大量存档数据可供参考,可应用于大规模研究。然而,扩增子测序确实有其局限性。首先它不能检测病毒,因为病毒缺乏类似于16S rDNA的保守基因;无法检测染色体基因组外的DNA,如质粒;在分析生物功能方面也存在不足;不完整的参考数据库可能会限制未知微生物的鉴定;菌群多样性的结果因不同可变区的选择而不同;仅基于单个基因的测序会忽略低丰度的目标微生物,导致假阴性;高宿主污染和低生物量样品出现目标产物放大质量欠佳的情况;该技术提供了较低的分辨率,因为单个基因的区域缺乏足够的序列可变性来区分物种,分辨率仅可以达到属级别。
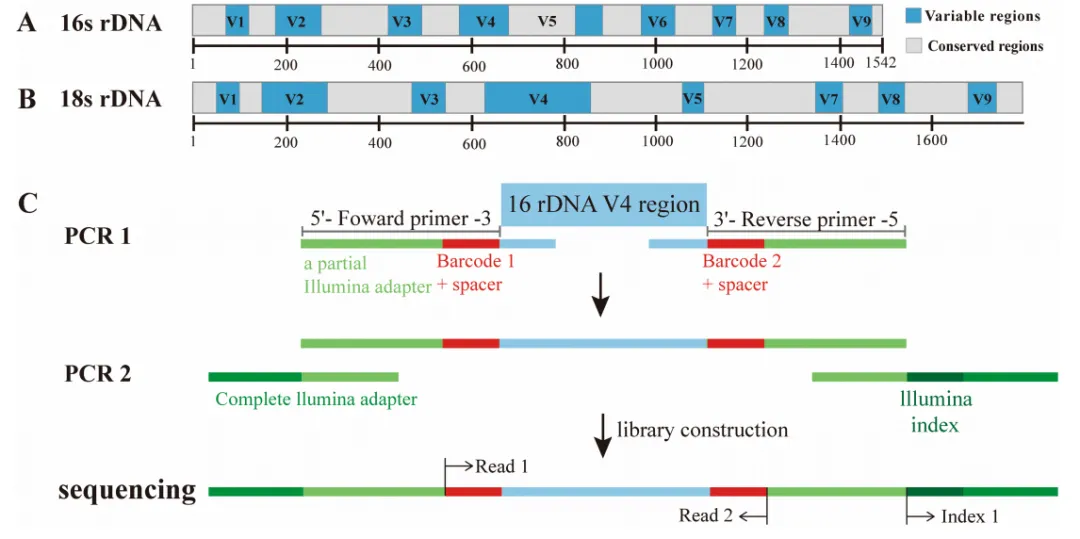
图2 扩增靶基因的结构及文库构建步骤
(A) 16S核糖体RNA (16S rRNA)长度约为1542bp,通常包括10个保守区和9个可变区(V1-V9),引物设计以保守区域为目标,放大一个或者多个可变区域(如V3或V3 - v4)。目标区域的选择取决于样本类型,并以已发表的文章为参考以确定最合适的v区。(B) 18S rDNA长约1500-2000bp,既有保守区,又有8个不同的可变区(V1-V9,无V6区),对于真菌来说,V1、V4、V5和V9可变区是最具区别性的。基于18S rDNA和ITS区域的扩增子测序是真菌鉴定最常用的方法。与ITS区域相比,18S rDNA基因在真菌群落中具有更高的稳定性。(C)文库构建原理。
02、宏基因组测序(mNGS)
宏基因组是特定环境下所有微生物(细菌、古生菌、真菌、病毒等)基因组的总和。利用高通量测序,将每个基因作为一个研究单元,研究人员可以研究物种多样性、群体结构、差异基因和功能基因。宏基因组的测序方法包括二代测序和三代测序。mNGS克服了传统纯培养方法的技术限制,使不可培养物种的研究成为可能。mNGS可以研究的样品类型包括土壤、水、粪便、大气样品、肠道内容物和植物内生菌。
与16S rDNA扩增子测序相比,mNGS具有多种优势。它可以在更精细的“物种”水平上进行物种识别,并且可以深入探索基因功能和代谢途径,这是16S测序分析无法做到的。然而mNGS也有一些弊端。宏基因组获得的基因组存在假阳性多、装配质量差、数据分析过程复杂、无法区分相似种、对菌株的鉴定能力非常有限等问题。在处理低生物量样品或宿主污染的样品时测序深度不足以预测单个物种的代谢途径,更侧重于整体途径的预测。
03、靶向测序(tNGS)
tNGS,也被称为病原体靶向测序,是一种利用靶向捕获的测序技术。该方法通过多重PCR富集特定致病微生物的基因组。随后进行高通量测序,并将获得的数据与参考数据库进行比较,以确定样品中是否存在致病微生物物种。多重PCR(mPCR)是一种在单一反应体系中特异性扩增两个或多个PCR片段的技术。具有同时放大不同模板的优点。传统PCR反应系统通常只能实现4-5个或最多30个反应,多重PCR采用数千甚至数万个重复的超高多重PCR技术,与NGS测序相结合,形成靶向tNGS技术。
tNGS在病原体检测领域具有若干优势。适用于住院患者病原体的初步筛查,提供常见病原体的全面覆盖,并提供粗略的定量信息。它不受人类基因组和背景菌群的影响,因为检测目标专门针对预先选择的致病性病原体,不包括人类基因片段和背景菌群的基因片段。该技术可以提高RNA病毒、真菌、细胞内细菌和吞噬病原体的检测能力。此外,tNGS可以直接扩增耐药和毒力相关基因,促进表型基因的检测。此外,通过添加新的引物对,可以根据临床需要随时添加新的靶点。与mNGS相比,tNGS在病原体检测方面具有更高的准确性和灵敏度。
然而,tNGS技术有一些局限性。它是一种较新的临床应用,目前仍处于快速推广阶段。该技术在识别新的病原体、检测某些病毒和检测罕见标本的能力方面面临挑战。致病微生物序列不完整或数据库中缺乏全面的序列会使鉴定少数病原体变得困难。
01、2bRAD-M简化微生物组技术
2bRAD-M简化微生物组技术是一种高度简化的微生物组测序技术。该技术利用IIB型限制性内切酶对基因组进行酶切,产生等长标签。以BcgI酶为例,它产生长度为32个碱基对(bp)的等长标签,每个基因组的标签数量约为3010个,其中39.7%为unique标签。随后酶切标签经过接头连接和片段扩增,进行文库构建和测序。测序数据与2bRAD特异性标签数据库(2b-Tag-DB)进行比对进行定性。2b-Tag-DB是一个由26万微生物基因组特异性标签组成的数据库,包括254,090种细菌、982种真菌和4316种古细菌。2bRAD-M简化微生物组技术能够以最高的准确性和灵敏度处理高降解、低生物量和高宿主污染的样品,同时识别细菌、真菌和古细菌,是微生物组分析的可靠方法(见图3)。
2bRAD-M简化微生物组技术在微生物测序方面有几个优势。具有很高的技术重复性,三个重复的平均L2相似度为95.4%。还具有很高的灵敏度,在1pg DNA起始量的样品中L2相似性也达到83.5%。另外,该技术有效地解决了三个关键挑战:高降解、低生物量和高宿主污染的样品,平均L2相似度分别为89.6%、84.6%和88.9%。2bRAD-M简化微生物组技术另外一个显著优点是允许在一个实验中进行多个分析,除了传统的微生物多样性分析外,它还可以分析宿主单核苷酸多态性(SNP)。这扩大了分析的范围,包括人类遗传分析,如亲缘关系分析、群体遗传结构分析和选择性消除分析。此外,2bRAD-M简化微生物组技术可以联合分析微生物多样性和宿主SNPs进行全基因组关联研究(GWAS),但宿主SNPs的数量可能有限。
与16S rDNA扩增子测序技术相比,2bRAD-M简化微生物组技术提供了更高的物种分辨率,能够识别”物种“水平的差异。它可以在一次实验中同时检测细菌、真菌和古细菌。该技术具有更高的准确性和灵敏度,因为它可以捕获整个基因组范围的信息,而16S rDNA扩增子测序则专注于基因的特定区域。
与mNGS相比,2bRAD-M简化微生物组技术的优势在于对DNA质量的要求较低,可以处理低生物量或严重降解的样品。此外,2bRAD-M简化微生物组技术可以有效地处理高宿主污染的样品,如组织、血液、体液和拭子。然而,2bRAD-M简化微生物组技术的一个局限性是它不能检测病毒。
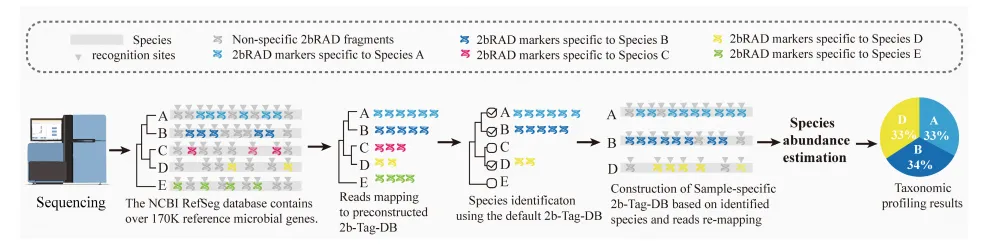
图3 2bRAD-M简化微生物组技术的流程
02、MobiMicrobe高通量微生物单细胞测序技术
单细胞技术是生物学研究的前沿领域。五种主要的单细胞分离技术包括荧光激活细胞分选(FACS)、激光捕获显微解剖、微操作、有限稀释和微流控技术,其中微流控技术因其巨大的潜力而备受瞩目,尤其是其能够实现每秒数千个单细胞的高通量分选能力。MobiMicrobe技术是一种高通量微生物检测方法,该技术能够获取单个微生物并组装其基因组,其中革兰氏阳性菌的基因组覆盖率为17%至25%,革兰氏阴性菌为8%至9%。特别值得一提的是,仅需20个Single-Amplified Genomes(SAGs)即可组装出完整的基因组。这项技术提供了多种应用,包括实现菌株水平的分辨率和发现难以培养或未知的新物种。
MobiMicrobe测序技术的工作原理基于微流体技术,将单个微生物及其裂解物封装在液滴中。随后,微生物被裂解,释放出DNA。扩增试剂被加入到每个液滴中,使得单个微生物的单细胞基因组得以扩增。接着,这些液滴与含有试剂的液滴结合,实现扩增DNA的片段化,并添加Nextera适配器。同时,条形码引物在单独的液滴中被加入,并利用聚合酶链反应(PCR)将这些条形码引物连接到片段化的DNA分子上。液滴破碎后,携带条形码的DNA片段被收集并进行测序。通过分析和比较每个单细胞的基因组特征,MobiMicrobe能够识别样本中同一物种的单细胞微生物。这些被鉴定的细胞随后被组合在一起,以物种水平组装出一个参考基因组。此外,通过将单个微生物基因组与参考基因组进行比较,可以区分来自不同菌株的单细胞微生物,并促进基因组组装(见图4)。
MobiMicrobe在微生物基因组学领域具有多项优势。首先,它消除了预培养细菌的需要,使得在一次测序中获得数千个个体微生物的基因组信息成为可能。由此产生的基因组组装质量高,可与通过分离培养获得的基因组相媲美,达到“金标准”水平。此外,MobiMicrobe支持功能基因和代谢途径的研究,为理解微生物功能提供了深刻见解。MobiMicrobe的一个关键优势在于其能够在菌株水平上精确解析基因组,从而发现传统培养方法可能遗漏的新物种和菌株。这解决了宏基因组方法的一个主要局限性,即在物种和菌株水平鉴定上的挑战。此外,MobiMicrobe有助于深入探索单细胞信息,使得菌株间的水平基因转移(HGT)分析成为可能。值得注意的是,同一属细菌之间的HGT比不同属之间的转移更为常见。该技术还可以在菌株水平上对宿主-噬菌体关联进行研究。尽管如此,MobiMicrobe也存在一些局限性。与宏基因组方法相比,它显示出较低的变形菌门和拟杆菌门细菌丰度。此外,革兰氏阴性菌的基因组覆盖率普遍低于革兰氏阳性菌,这可能与所选的裂解方法有关(见表1)

图4 基因组组装和菌株水平的比较分析
03、2bRAD-M简化微生物组技术和MobiMicrobe在微生物组研究中的潜力和缺陷
人类微生物组在疾病的发展中扮演着关键角色,深入理解微生物与宿主细胞间的相互作用对于疾病的诊断、治疗和预防至关重要。当前,探究微生物的功能机制已成为研究领域的热点。2022年,两项革命性技术—2bRAD-M简化微生物组技术和MobiMicrobe—被引入微生物组研究领域,它们克服了传统测序方法的局限,为微生物研究开辟了新天地。2bRAD-M简化微生物组技术使得跨批次数据的合并分析成为可能,同时检测细菌、真菌和古细菌,并分析宿主的单核苷酸多态性(SNP)。它实现了物种级别的分辨率,有效应对低生物量、高降解或高宿主污染的样本挑战。而MobiMicrobe技术则提供了与“金标准”相媲美的可靠基因组组装,能够精确分析菌株水平的基因组,并揭示新的未培养菌株。此外,它还有助于探究微生物群落内部菌株间的相互作用。
尽管如此,这些技术在未来发展中仍面临一些限制和挑战。例如,2bRAD-M简化微生物组技术无法检测基因组小而短的病毒。对于MobiMicrobe技术而言,与宏基因组方法相比,其在变形菌门和拟杆菌门细菌的丰度上相对较低,且革兰氏阴性菌的基因组覆盖率通常低于革兰氏阳性菌,这可能受到裂解方法的影响。
高通量测序技术的持续进步有望对微生物学领域产生深远的影响。首先,测序速度更快、成本更低,同时数据更准确。其次,使探索微生物群落结构和相互作用成为可能,从而揭示微生物间的共生关系和生态功能。此外,这些技术将有助于阐明与代谢、抗生素耐药性和毒力因子相关的微生物功能特征。第三,高通量测序技术将推动微生物与宿主相互作用的研究,通过深入研究微生物群与宿主相互作用的复杂网络,我们能够获得微生物如何影响宿主免疫系统、代谢调节和疾病发展的宝贵见解。这些知识对于理解微生物对人类健康和疾病的影响至关重要。最后,这些高通量技术将促进微生物精确诊断的发展,包括微生物标记物的识别,以及治疗靶点和策略的发现,为健康管理和疾病预防提供个性化的微生物组干预策略。
【参考文献】
Yi, Xin et al. “Unravelling the enigma of the human microbiome: Evolution and selection of sequencing technologies.” Microbial biotechnology vol. 17,1 (2024): e14364. doi:10.1111/1751-7915.14364






