EzBioCloud 介绍
对于微生物方向的老师,科研工作中是离不开EzBioCloud数据库的。这是一个由ChunLab维护的专门针对细菌、古菌16S rRNA基因的数据库,但与Greengenes数据库不同的是,该数据库以可培养的细菌、古菌16S rRNA基因序列为主。我们可以通过EzBioCloud数据库的Identify功能确定某一微生物的近缘可培养/模式种。当然,如果我们想要获得某一个属的所有可培养种的16S序列,EzBioCloud也是很好的选择之一。
我们可以冲一杯咖啡,然后一层一层的点进去下载(编码 U+2615)
脚本逻辑
但是,生信人普遍存在的一个特点就是“懒”,换言之,逻辑性的工作留给自己,重复性的工作留给机器,既然我们能逐个的下载,自然也可以教会机器帮我们逐个的下载。
首先捋一下脚本写作思路:
1. EzBioCloud数据库没有下载接口,且网页是异步加载,所以我们采用urllib+bs4的策略,抓取属内种的信息;
2. 抓取到菌株名和登录号以后就简单多了,直接交给biopython下载就好啦;
3. 当然,通过观察发现,EzBioCloud提供的登录号有些是全基因组序列,那我们就再加一步简单的过滤。
脚本介绍
明确思路以后,一顿劈里啪啦,脚本就出来了(编码 U+1f38a)

· -i 需要下载的属名,不区分大小写,但是请给定准确的属名(不能与-l参数共用);
· -l 需要下载的菌种列表,第一列为登录号,第二列为菌种名(不能与-i参数共用);
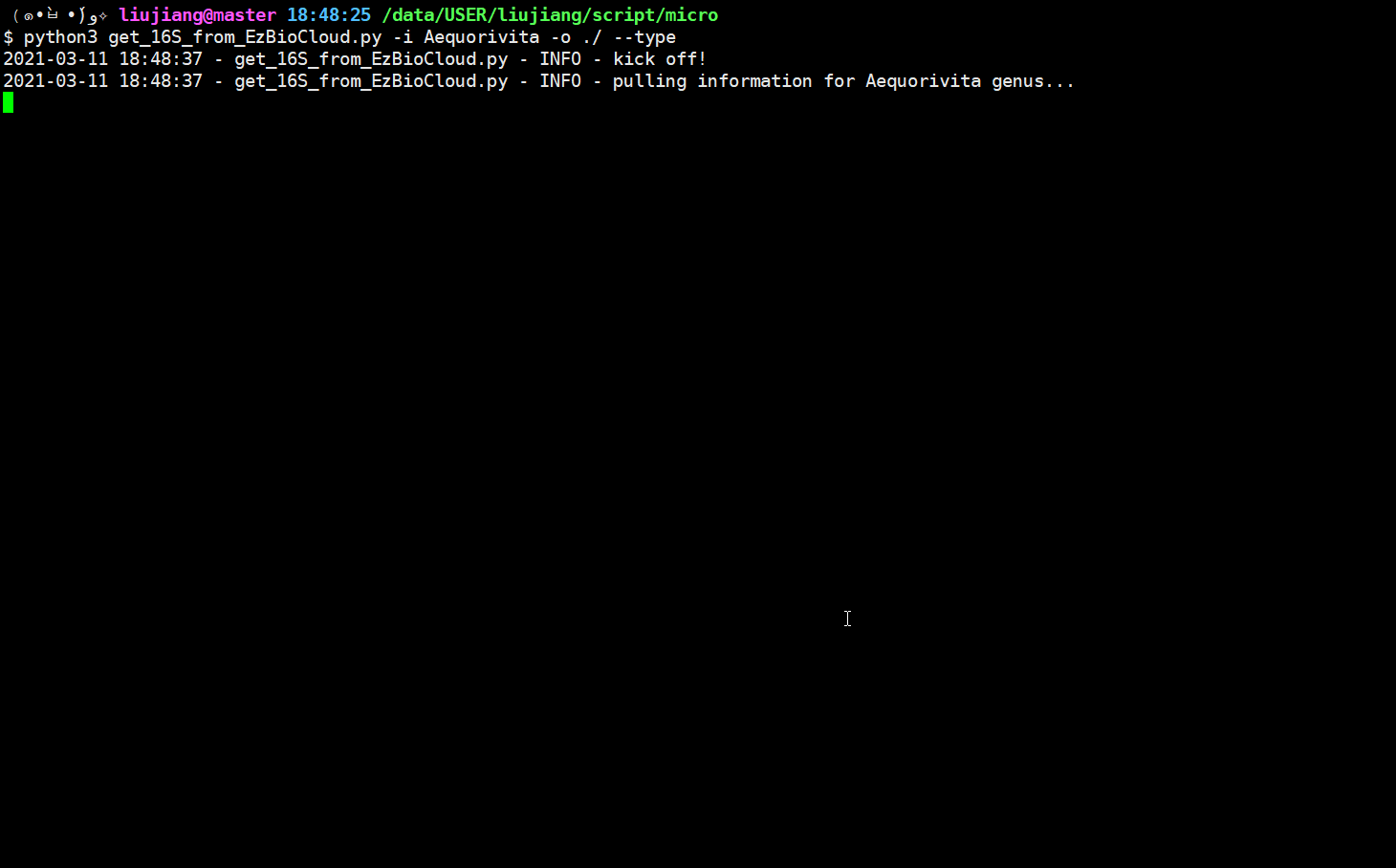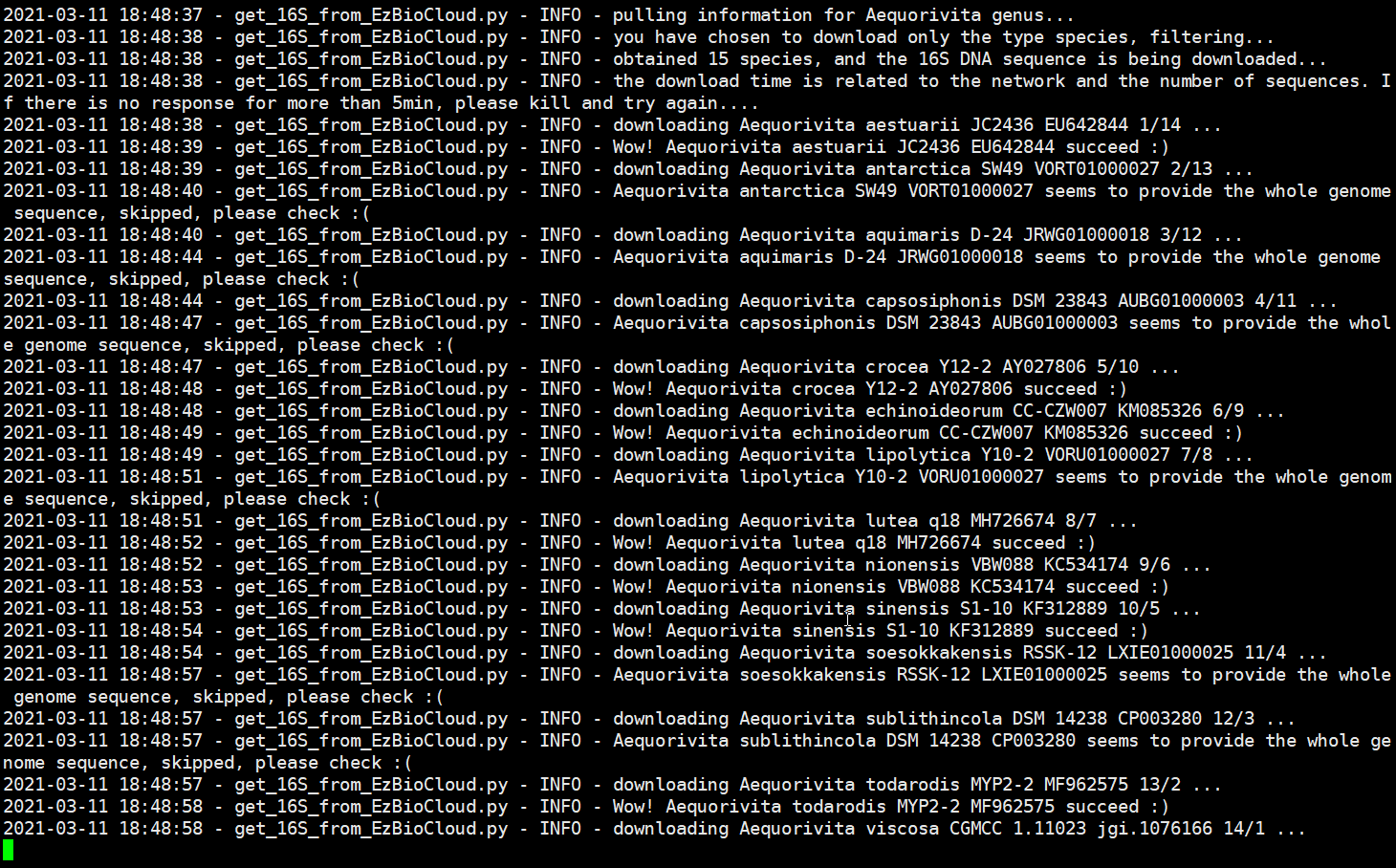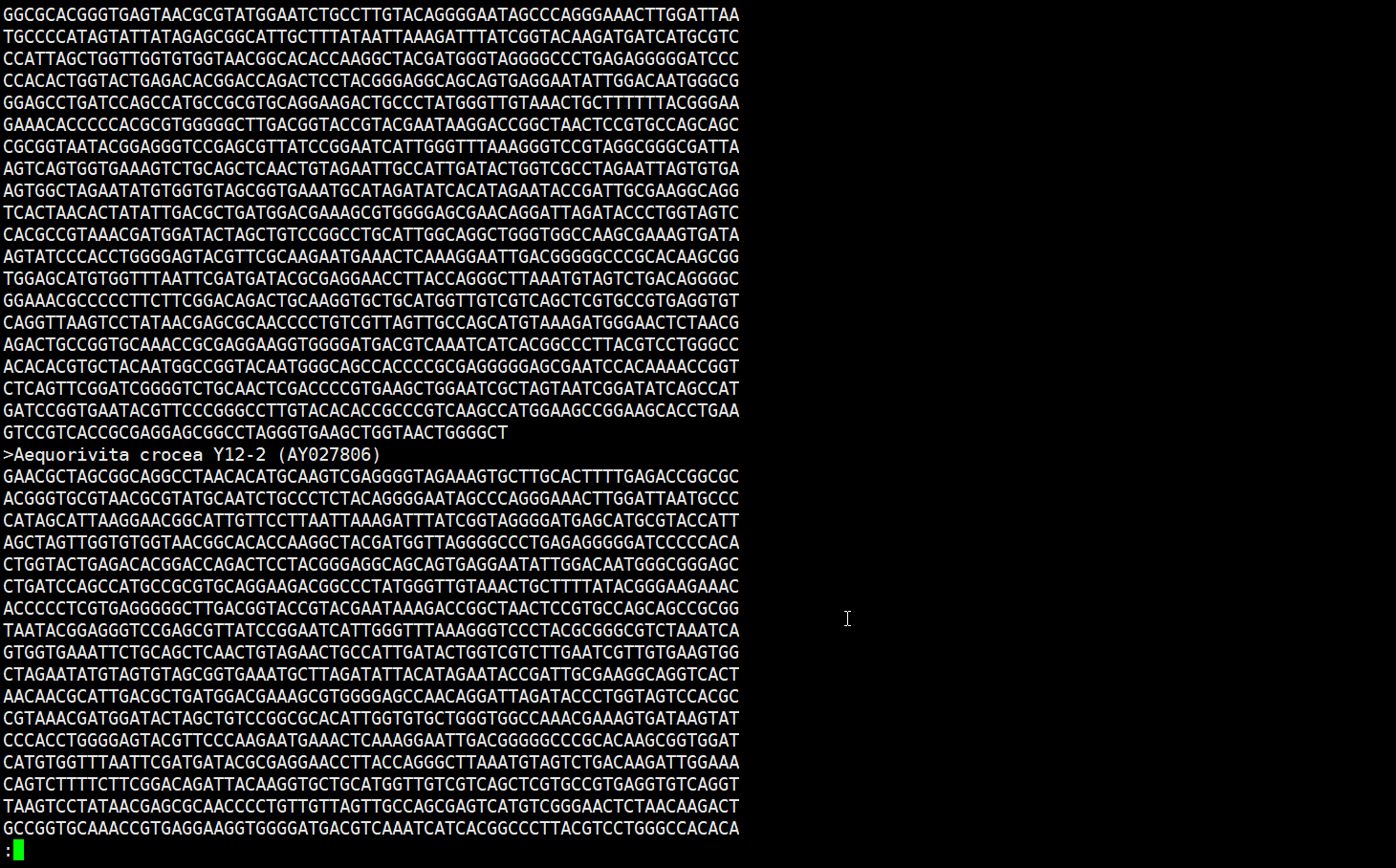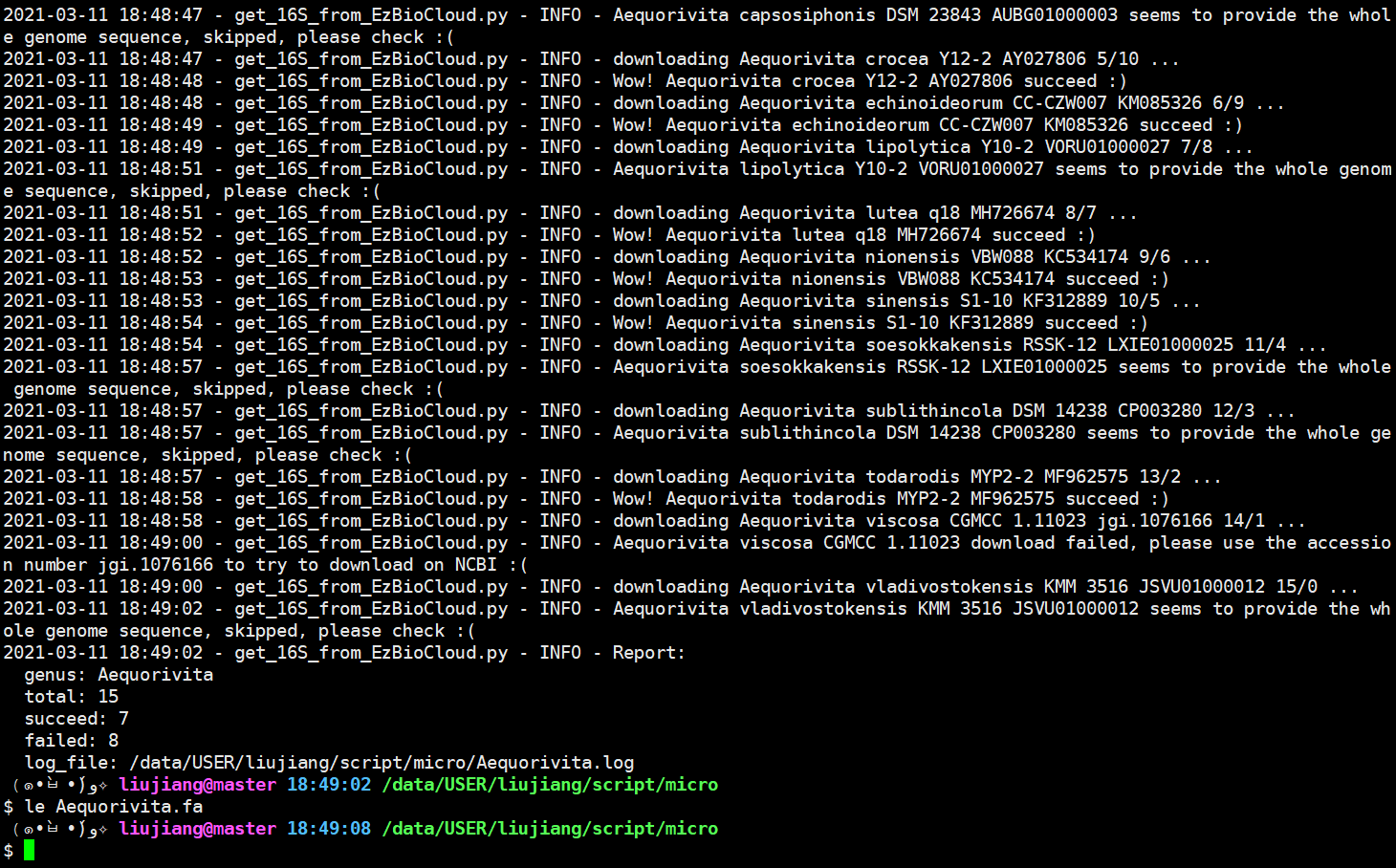
· -o 输出目录,将会在该目录中创建 *.fa *.log 和 *.xls 三个文件,分别记录下载序列、运行报告及菌种信息;
· --type 给定该参数时,脚本会对抓取到的菌种进行过滤,只保留模式种,该参数默认缺省。
对于新种鉴定工作,模式种更重要,所以脚本可选下载全部种或者只下载模式种;受限于网络问题,难免有下载失败的情况,所以脚本也可给定下载列表重新尝试下载。
脚本测试
对脚本进行了测试,似乎可以省不少的时间呢,这样我们就可以悠闲地喝咖啡啦!
最后给各位看官放上脚本下载地址:https://pan.baidu.com/s/18PLHcqAf3wK2uiY-AEjeFA,提取码: crpx。(可直接点击链接哦~)






