尽管在过去的几十年里,DNA测序成本大幅降低,但研究人员仍然面临着从三个方面考虑测序事宜:1)基因组覆盖度,即被测到的碱基占全基因组大小的比率;2)每个样本的测序深度,即每个碱基被测到的次数;3)测序的样本总数。
简化基因组测序(RAD-seq)的出现使得全基因组变异模式检测取得了突破进展,但是基因组大部分标记未获得是简化基因组的重要限制因素之一。
混池测序(Pool-seq)对于群体研究来说能够有效降低成本,但是,混池测序丢失了个体信息,无法进行个体层面的分析,也无法检测个体之间隐藏的信息。
有没有一种方案既能在群体水平对整个基因组进行研究,又能同时保留个体信息,而且其费用和RAD-seq、Pool-seq相当呢?
低深度重测序(lcWGS)正成为一种有效的替代方案,它借助概率统计策略,通过牺牲测序深度,以换取更大的基因组覆盖度和更大的样本量。那么低深度重测序可以分析什么类型的数据呢?什么样的实验设计可以获得最可靠的结果呢?今天带来低深度重测序群体研究之新手入门指南,希望能给大家带来帮助。

低深度重测序入门指南于2021年7月发表在Molecular Ecology,共包括四部分,详述如下。
一、 低深度重测序的成本是多少呢?
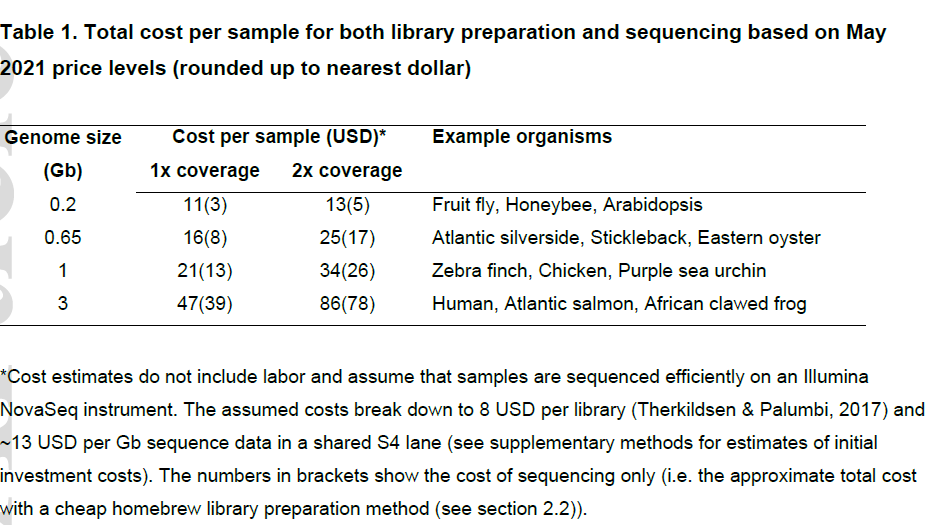
对于基因组较小的物种来说,低深度重测序可能比简化基因组(RAD-seq)方法更便宜,而且随着测序成本的继续下降,价格可能会进一步下降。另外,对非模式生物来说,低深度重测序的限制条件是参考基因组。如果没有参考基因组,常规的解决方案是选择近缘物种。三代测序技术的发展,使得可以较低的成本获得染色体水平的基因组。因此,如果没有参考基因组,基因组de novo对启动新的低深度重测序研究可能是有意义的。
表1 低深度重测序建库和测序总成本
二、 低深度重测序可以做哪些类型的分析呢?
低深度重测序的主要挑战是不能准确地推断个体基因型。因此,近年来发展了许多相关的分析工具,涵盖了绝大多数常见的群体类型。目前,低深度重测序分析中最广泛使用的软件是ANGSD,这是一个全面的软件包,包含了各种分析选项。当然,对于大多数类型的分析,都可以使用各种各样的替代软件。
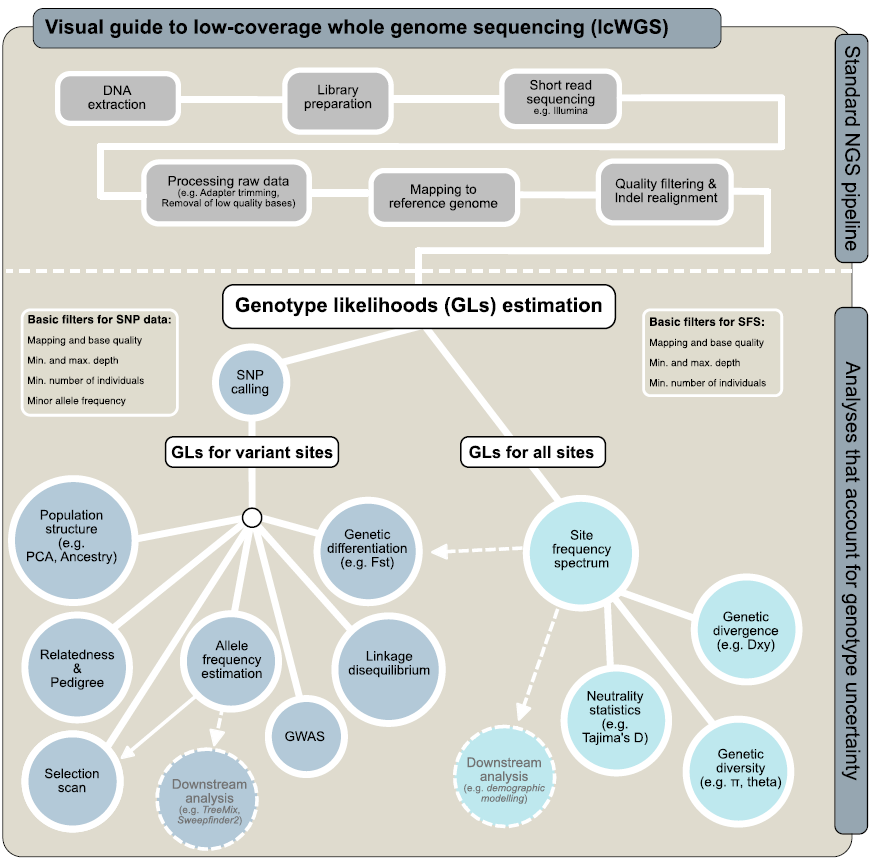
lcWGS流程图。上图:lcWGS预处理流程,和NGS流程非常类似。下图:lcWGS数据分析流程,它是使用基因型可能性来解释基因型不确定性的概率框架。从等位基因频率谱(site frequency spectrum,SFS)到多样性统计和FST,是ANGSD软件的分析流程。其他工具(如ATLAS)可以直接从GLs推断这些统计数据,而无需事先使用SFS。
三、 低深度重测序如何设计实验?
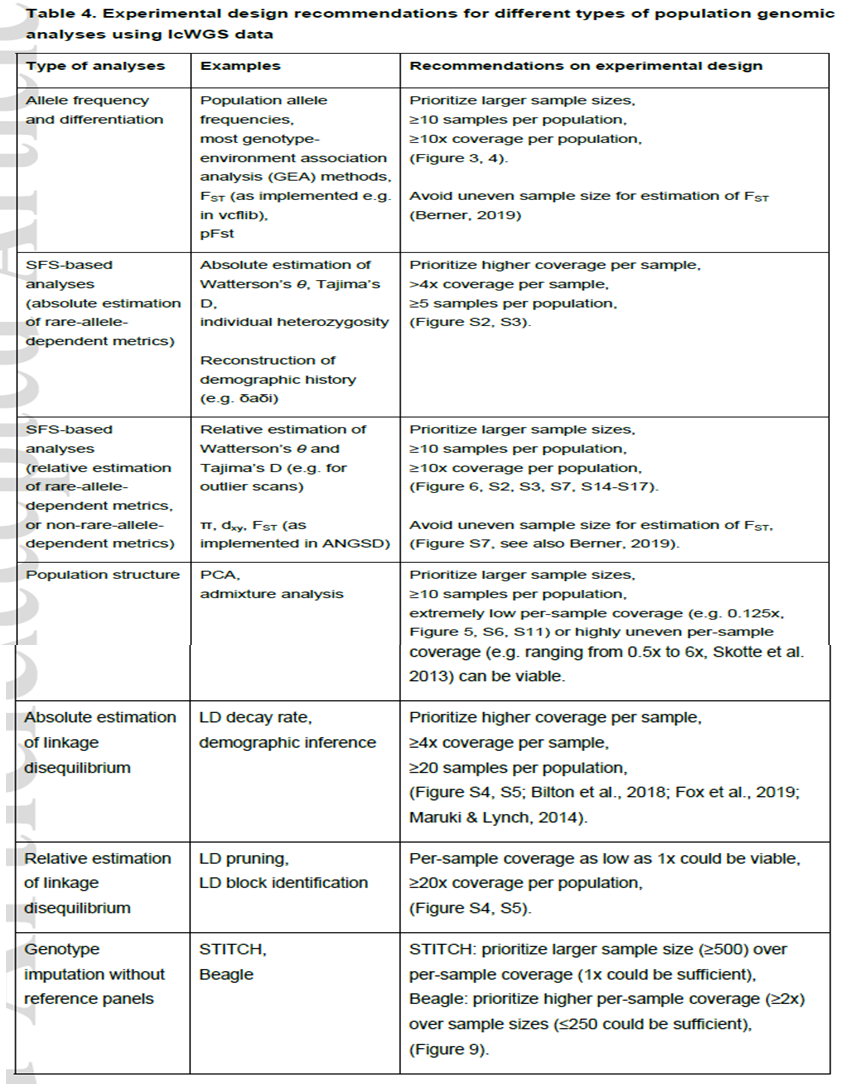
没有一套单一的低深度重测序实验设计适合所有研究目的。相反,最优设计取决于研究的目标、系统和预算。在给定预算的情况下,低深度重测序主要权衡的是样本数量和测序深度。例如等位基因频率估计、群体结构分析、群体间的遗传分化等,可以通过更多样本的测序来获得准确的结果;等位基因频率谱(SFS)、使用δaδi进行人口统计推断、Tajima’D或LD绝对值,需要考虑更高的测序深度。因此,研究人员必须仔细考虑哪种类型的分析对研究目标是最重要的,并找到适当的平衡。综合我们的和以前的研究结果,我们提供了一些低深度重测序实验设计的一般指导原则。如果要优化特定的实验设计,也可以采用已建好的模拟流程(https://github.com/therkildsen-lab/lcwgs-simulation)。
表2 采用低深度重测序进行不同类型群体分析的实验设计建议
四、 低深度重测序有哪些局限性?
尽管低深度重测序有许多优点,但这种数据类型在群体研究方面也有明显的局限性。
1) 不适合需要调用基因型的分析;
2) 缺乏用户友好的软件界面和文档;
3) 计算成本高;
4) 工具箱存在缺陷,如基因型判读模型的灵敏度、比对错误等;
5) 当不同数据库整合时,易受批次效应影响;
6) 在没有参考面板时不能准确分相,因此不能进行基于单倍型的分析;
7) 不适合小样本量或基因组非常大的场景。
参考文献
RN Lou, A Jacobs, A Wilder, et al. A beginner’s guide to low-coverage whole genome sequencing for population genomics. 2021, Molecular Ecology, DOI:10.22541/au.160689616.68843086/v4
小欧推荐
低深度重测序将给定数量的测序数据量分散到更多的样本中,虽然每个样本深度降低,但是仍然可以提高大多数群体类型推断的准确性。虽然低深度重测序还存在一些限制,但这种方法比简化基因组和混池测序提供了许多优势,可以广泛应用。另外,低深度重测序的多功能分析工具箱正在迅速扩大,这使得它成为分子生态学、保护和进化生物学研究的一个越来越有前景的方法。






