大家还记得上次分享的:《科研干货 | BRIG绘制原核生物比较基因组圈图》文章么?文章发布后小编收到了很多老师的咨询,希望我们再来篇基因组共线性分析软件的文章。
不怕小编没存货,就怕不知道老师们想要什么。
今天就给各位老师介绍一款强大的共线性分析软件:Mauve[1]。Mauve可用于构建多个小型基因组的比对,可以解释进化关系,也能够快速有效分析基因组之间有无大片段序列重排现象,及基因组间共线性是否良好,以及是否存在局部共线区(Locally Collinear Blocks, LCBs),这些分析都为比较基因组学的研究和全基因组进化动力学的研究提供了基础。
先甩上官网链接:
http://darlinglab.org/mauve/mauve.html
先来康康Mauve做的图
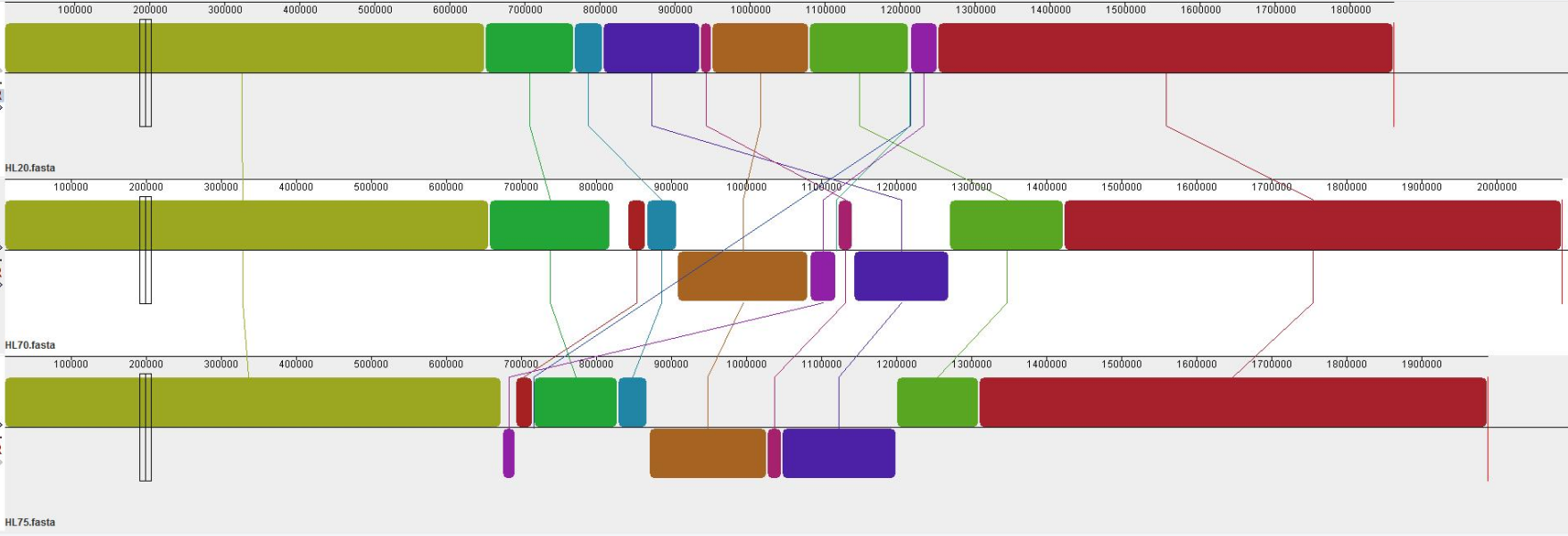
一、软件下载
安装Mauve需要先安装Java,并配置到环境变量中(在上次的的BRIG软件操作中推文已分享,在这里就不赘述啦~)
Java下载地址:
https://www.oracle.com/java/technologies/javase-downloads.html
https://www.java.com/zh-CN/download/manual.jsp
Mauve软件下载地址:
http://darlinglab.org/mauve/user-guide/installing.html
点击mauve_installer_20150226.exe,安装即可
运行界面如下图所示,就说明安装成功啦:
二、软件操作
2.1安装完成后,准备菌株.fasta或者.Genbank文件
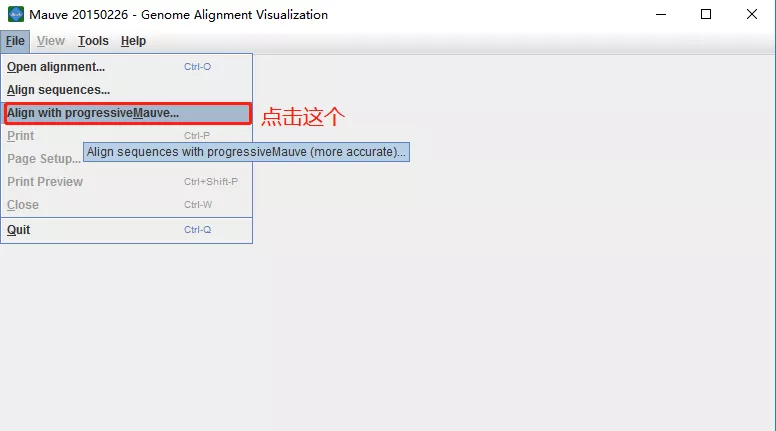
打开Mauve软件,在这里选择使用Align with progressiveMauve...
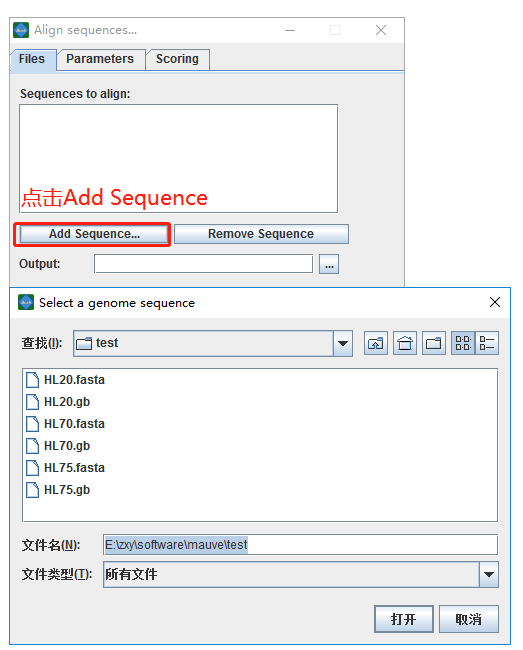
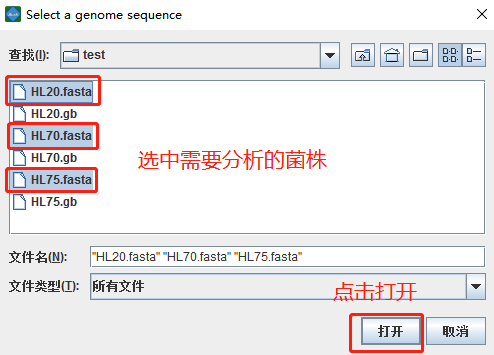
2.2 点击Add Sequence,在这里我下载的是NCBI上lactobacillus gasseri的三株菌的fasta文件,上传后点击打开即可。
2.3 点击打开出现下图界面,输入文件位置及名字,点击Align即可
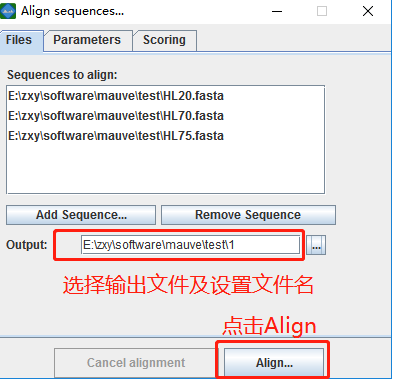
2.4 运行过程中会出现一下运行界面,运行需要等一会
运行结束后图就出来啦~~~
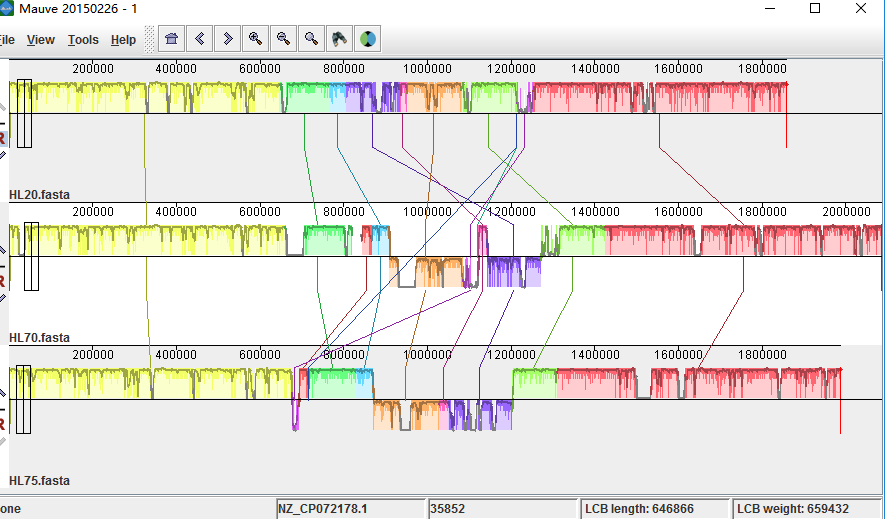
先要修改颜色啥的,在菜单栏里View里下选择展示的效果。Mauve多基因组比对可以导出共同的基因(基因重排,缺失,结构,同源基因以及每个基因组的独特基因),相关的功能都在Tools下的Export见详情。
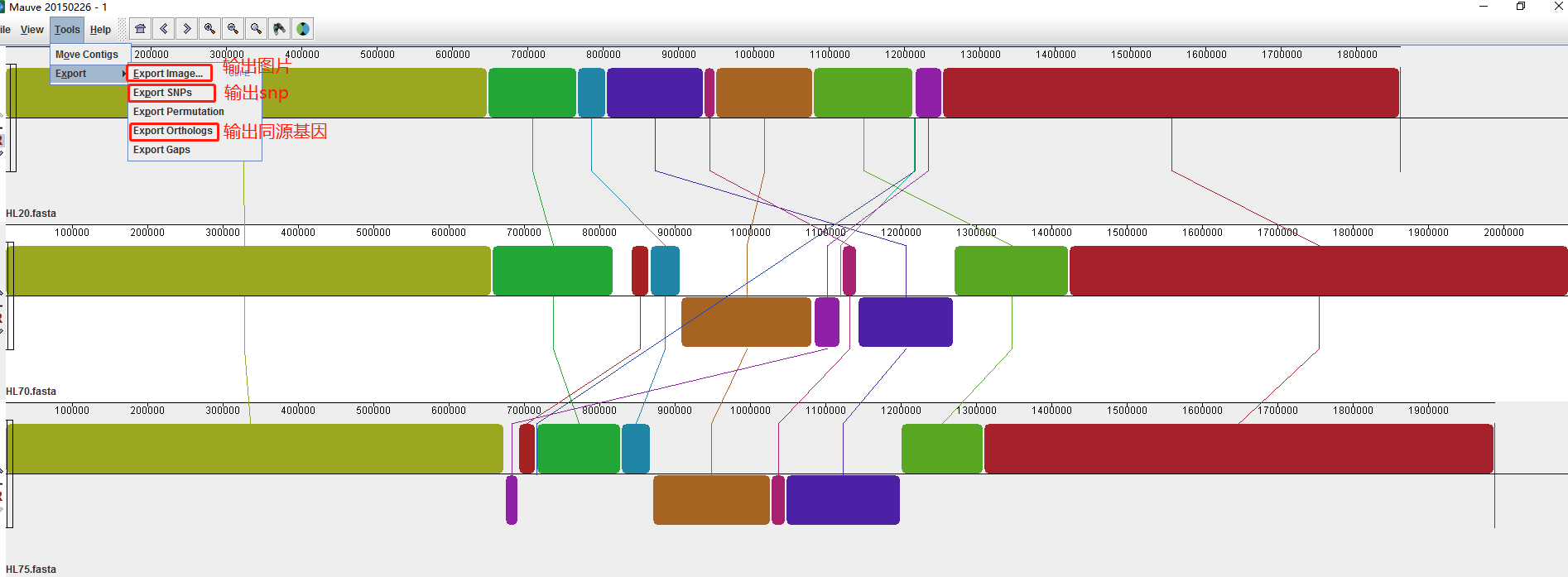
好啦今天的分享就到这里啦。心动不如行动,各位赶紧试试吧!
参考文献
[1]Darling A, Mau B, Blattner F R, et al. Mauve: Multiple Alignment of Conserved Genomic Sequence With Rearrangements[J]. Genome Research, 2004, 14(7):1394-1403.
精彩回顾






