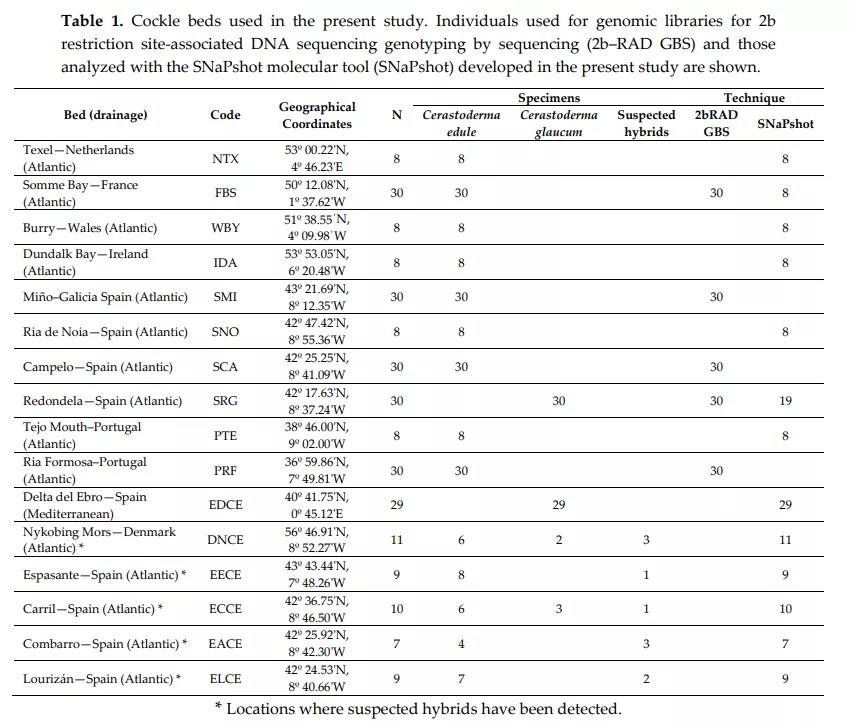
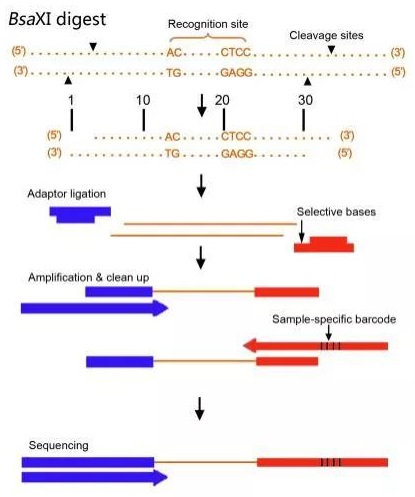
论文名称:A Useful SNP Panel to Distinguish Two Cockle Species, Cerastoderma edule and C. glaucum, Co-Occurring in Some European Beds, and Their Putative Hybrids 发表期刊:Genes 影响因子:3.331 涉及的欧易生物服务产品:2b-RAD 研究背景 蛤是非常珍贵的软体动物,在欧洲有两种不容易被区分的贝类,一种是可食用的Cerastoderma edule(以下简称C. edule),另一种是不可食用的Cerastoderma glaucum(以下简称C. glaucum)。它们对寄生虫Martelia cochillia表现出不同的抗性,C. edule对这种寄生虫十分敏感,而C. glaucum似乎具有抵抗力。虽然潜在的原因尚不清楚,但是,通过自然或人工的方式,利用杂交完成基因渗入,使可食用的C. edule获得对Martelia cochillia的抵抗力,并研究这种杂交物种的生存与繁殖是很有意义的。 Cerastoderma edule 图片来源:guiamarina.com 研究内容 C. edule和C. glaucum很难一眼就分辨出来,尤其是在它们还小的时候。但是通过对它们形态特征的研究和积累的经验,还是可以区分的。首先对16个分布广泛的采集点进行190个纯种样品的采集,然后对其中150个样品,使用2b-RAD筛选出9个SNP;其次使用SNaPshot进行验证,获得7个SNP;最后再用SNaPshot方法检测两个物种是否为杂交物种。虽然这些杂交物种被证明是假阳性,但我们不能排除杂交可能发生和可行的事实;并且,文章中筛选出7个SNP来区别C. edule和C. glaucum,建立了区分这两种贝类及其杂交种的SNP面板。对研究marteiliosis的抗性也有重要帮助。 研究结果 1. 采用2b-RAD技术 利用2b-RAD技术对2017-2018年收集的30个C. glaucum个体和120个C. edule个体进行基因分型,采用IIb限制性内切酶AlfI,Illumina 测序平台进行测序。按质量值(Phred值> 30)进行过滤,使用Stacks 2.0进行比对。最终获得了9个SNP位点。 2. 采用SNaPshot筛选对选定的SNP进行验证 该方法基于两步扩增反应方案对SNP进行荧光检测。第一步是对包含所选SNP的区域进行PCR扩增(使用外部引物),第二步是对相邻SNP引物(即内部引物)进行碱基测序反应,内部引物设计长度差异通过在3 '末端添加CAGT序列来进行测序。 根据上一步筛选出的SNP位点上、下游150 bp的基因组序列,利用Primer3软件设计外部引物。Amplitaq GoldTM DNA聚合酶进行扩增,只选择在琼脂糖凝胶中条带大小符合要求并单一完整的样品在ABI 3730xl 中进行检测,内部引物可用于基因分型。这些序列使用BLASTn默认参数进行注释。最终筛选出7个SNP位点。 3. 疑似杂交物种的鉴定 选46个样品(包括31个纯合C. edule,5个纯合C. glaucum,10个疑似的杂交物种)进行SNaPshot验证,结果与ITS方法相反,全部为纯合子。虽然这些杂交物种被证明是假阳性,但我们不能排除杂交可能发生的事实。 研究结论 1. 经过质量过滤,平均每个标本保留2768161条reads。两个物种共识别了13715个SNP, 2057个诊断型位点和11,658个限制型位点。在限制型位点中,1658例为C. edule(1190例的MAF≥0.05;占71.8%),10,000例为C. glaucum(3028例的MAF≥0.05;占30.3%)。这些研究表明,C. glaucum的遗传多样性远高于C. edule。 2. 作者用他们开发的PANL鉴定了10个可能的杂交个体,结果却是纯合的,并且在9个SNP位点上都是纯合的,但是以前的ITS工具鉴定这些个体是杂交个体,所以得出了矛盾的结果。虽然这些杂交物种被证明是假阳性,但我们不能排除杂交可能发生的事实。 3. 使用两种技术(即2b-RAD和SNaPshot)分析的个体中获得的基因型是相同的。这9个标记的序列证实,这些标记在两个物种之间具有单一的诊断差异(GenBank登录号:MN178488-MN178496)。最终开发出了针对这个品种的7个位点的特异分子标记,构建了PANL,为之后的研究提供了方法思路。 青岛欧易 2b-RAD简化基因组测序 技术简介 2b-RAD技术使用IIB型限制性核酸内切酶(如BsaXI)对基因组DNA进行酶切,切割位点位于识别位点的左右两侧,酶切后产生等长的33 bp的标签,这些标签经过富集后用于高通量测序,通过生物信息学分析实现全基因组范围高通量SNP筛查和分型分析。 2b-RAD技术流程 技术特点 1. 适用于降解样品、痕量样品(ng级DNA); 2. 技术重复性好,标签长度一致、测序深度均一,SNP准确性和利用率高。 IIB型内切酶酶切标签图解 (“红色碱基”为酶切识别位点,左右两侧切割产生粘性末端,切割位点与识别位点的相对距离是固定的,基因组各位置切下来的标签等长) 青岛欧易 青岛欧易生物科技有限公司多年来,立足生命科学,开发特色技术,为农、林、牧、渔等领域科学家提供遗传及分子育种方向的高端特色技术服务。目前在简化基因组测序研究已有了成熟的技术方法,欢迎各位老师前来咨询噢~ END 青岛欧易 撰文 本文系青岛欧易原创 转载请注明文本转自青岛欧易