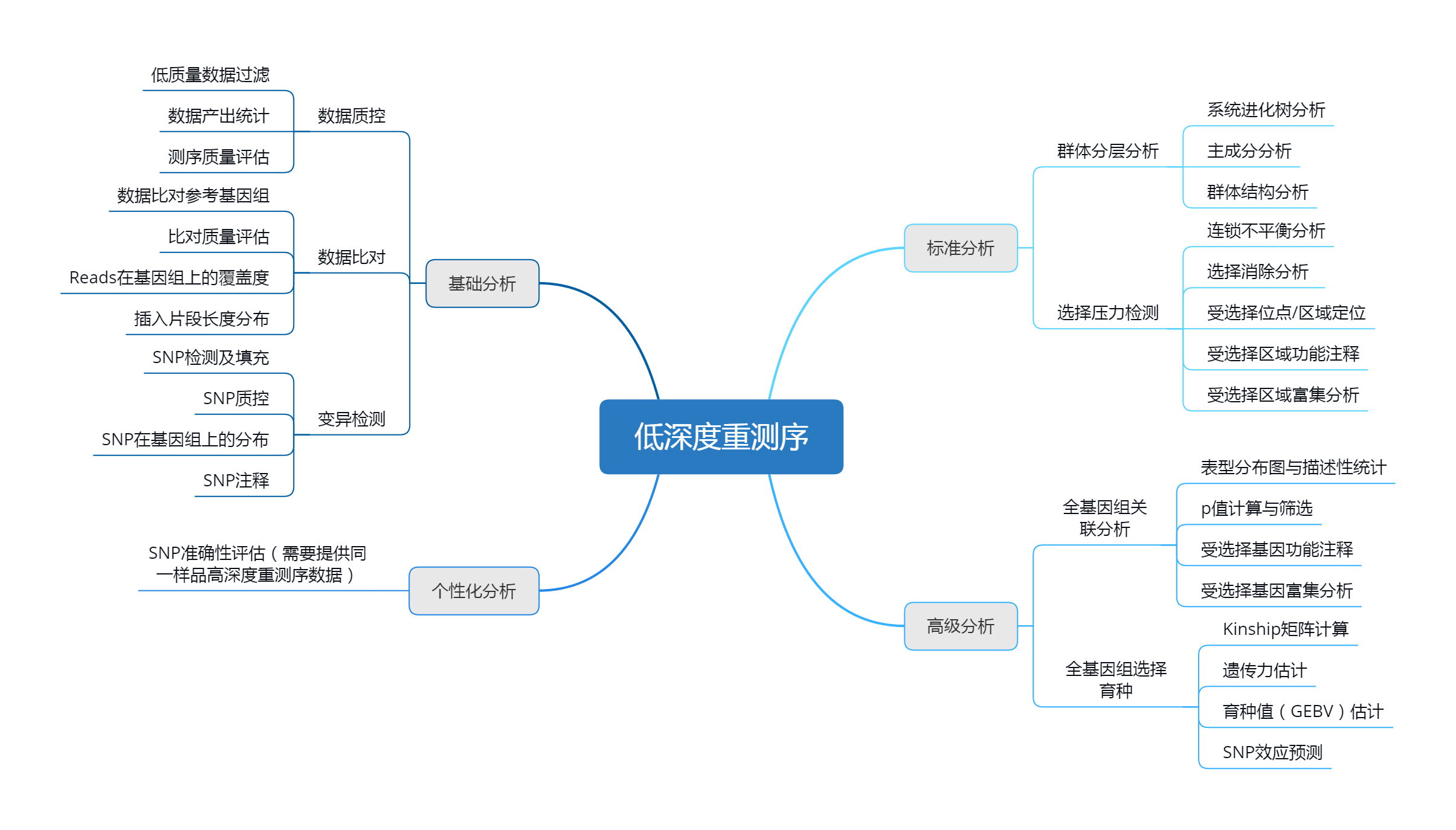
低深度重测序(LcWGS)
全基因组低深度重测序(low-coverage whole-genome sequencing, LcWGS)是继简化基因组测序之后新一代低测序成本的标记开发方法。一般认为其测序深度在1×以下,该策略首先对群体中所有个体进行全基因组低深度重测序和变异查找,之后根据SNP位点间的连锁不平衡(linkage disequilibrium, LD)对缺失基因型进行推断和基因型填充(genotypeimputation),最终获取大规模样本全基因组水平的高密度遗传标记。
基因型填充是根据参考面板(reference panels)中的单体型和基因型对目标样本中缺失基因型的估计填充过程,可有效增加单核苷酸多态性(SNPs)密度,其示意图如下:
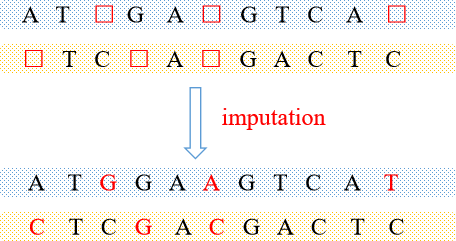
△ S1和S2代表样品,£代表缺失基因型,A、T、C、G代表已知基因型
技术比较
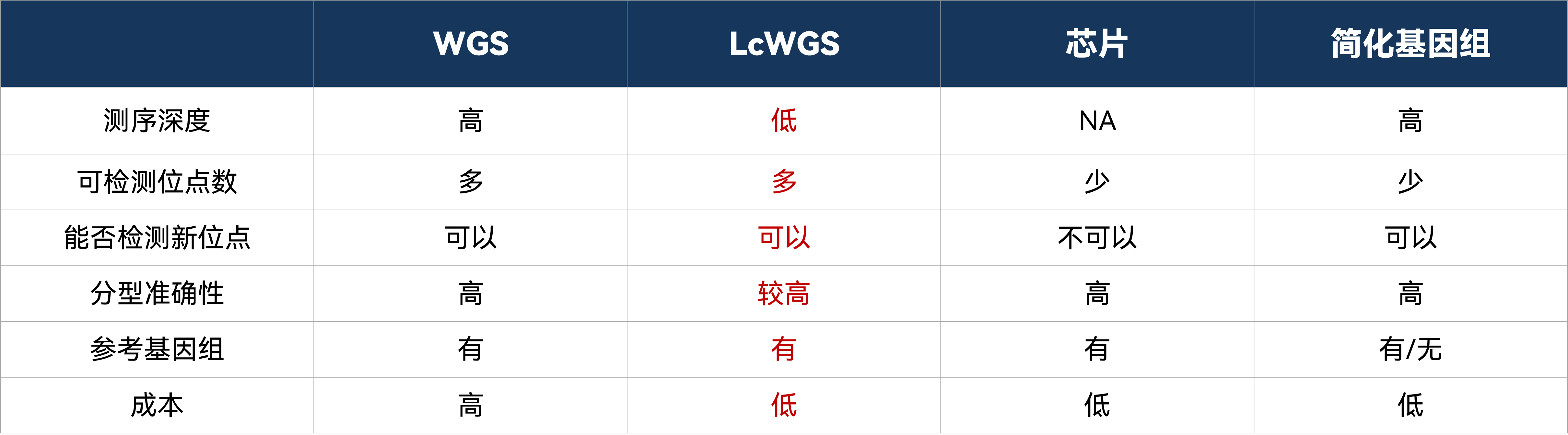
目前高通量SNP检测方法主要包括全基因组重测序(WGS)、SNP芯片、简化基因组测序等。其中全基因组重测序理论上能检测基因组范围的所有变异位点,但是测序成本高昂;SNP芯片检测标记数量有限、只能检测已有突变,目前商业化的SNP芯片只包含部分物种;简化基因组测序技术相较于SNP芯片,检测位点更多且不受物种限制,但只能检测基因组2%左右的变异位点。
低深度重测序技术可以减少测序数据量降低测序成本,通过基因型填充得到全基因组范围内的高密度SNP,对于全基因组关联分析(GWAS)和基因组选择(GS)而言,低深度重测序技术可以满足它们高SNP密度和大样本量的需求,性价比更高。
全基因组重测序、SNP芯片、简化基因组测序和低深度重测序技术比较如下:
技术特色
物种丰富
18物种已有panel,其他物种可定制panel。参考面板库链接为:
http://gong_lab.hzau.edu.cn/Plant_imputeD-B/#!/
http://gong_lab.hzau.edu.cn/Animal_ImputeDB/#!/

△已有panel物种图
基因型填充准确度高
评估了猪( P i g )、牛( C a t t l e )、绵羊( S h e e p )、水稻(Rice)、油菜(Oilseed rape)5个物种SNP填充的准确率,随着测序深度的增加,SNP填充准确率提高,在1X测序深度时,平均准确率达95.9%,其中在0.3×测序深度下,水稻基因型填充的准确率达到98.2%,绵羊基因型填充的准确率达到97.9%。
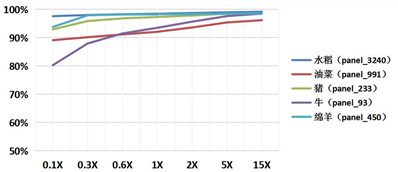
△ 基因型填充准确率图
横坐标代表不同测序深度,纵坐标代表基因型填充的准确率,
不同颜色折线代表不同物种,“panel_数字”数字代表该物种panel中包含的个体数
辅助分子育种
助力全基因组关联分析和全基因组选择研究
服务优势
分析流程