此外,10x还专门提供了最多480个基因的探针全定制服务,可满足Xenium在非人和小鼠的物种中进行基因表达水平的检测。虽然10x为定制探针的设计提供了便捷的软件Xenium Panel Designer,但对于没有相关经验的小伙伴来说可能还需要花费一定的时间进行学习和摸索。为此,我们梳理了Xenium预制panel增加探针定制的标准设计流程和注意事项,便于有需要的小伙伴快速上手,把Xenium的高端检测技术运用到科研实践中。
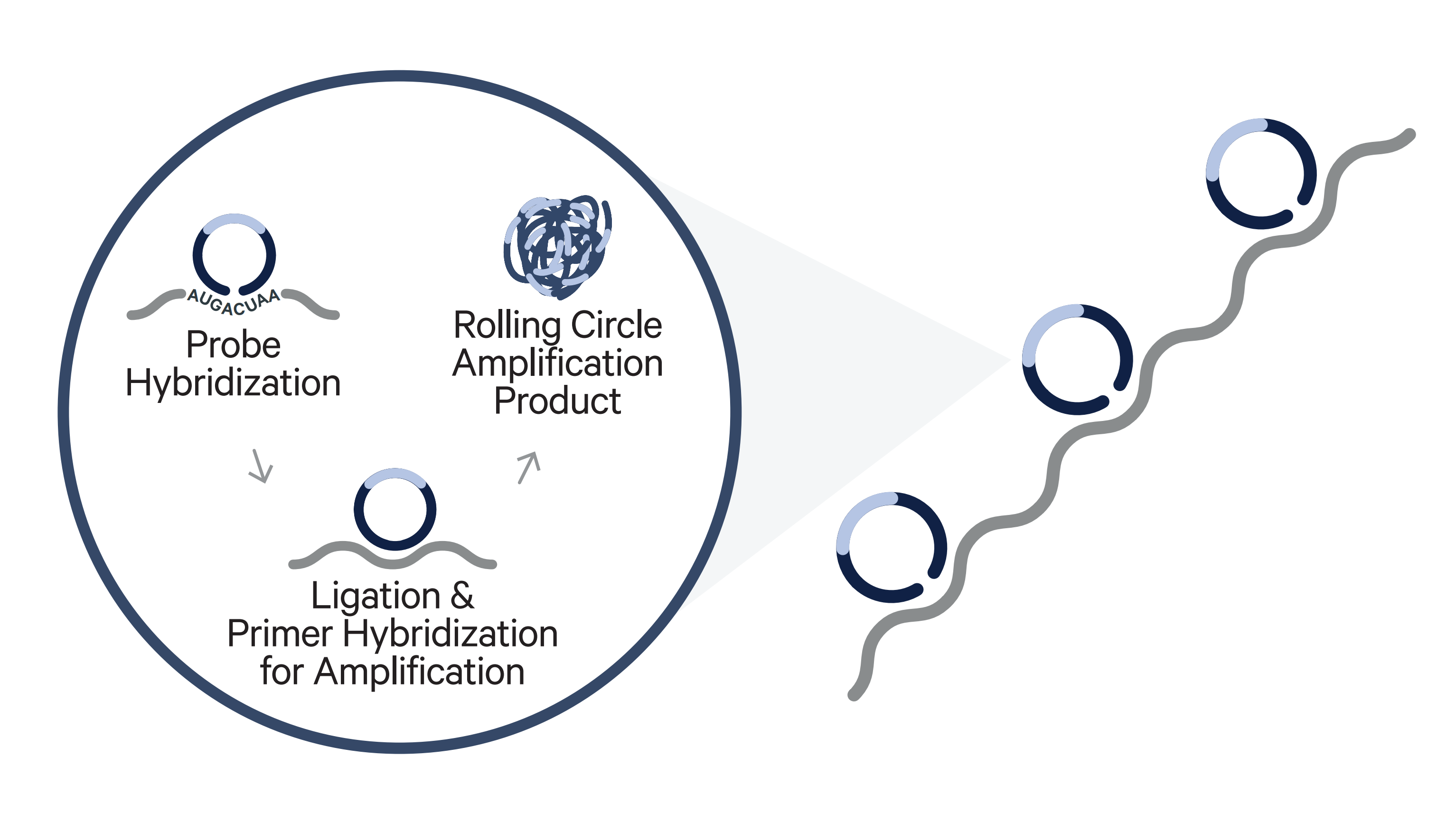
图1 | Xenium探针示意图(图片来自10x Genomics)
针对客户不同的探针定制需求,10x Genomics提供了两种可选流程:
1)标准定制流程(Standard workflow):客户利用Xenium Panel Designer软件完成探针设计,提交定制需求,获得定制ID,然后联系对应服务商或经销商进行付费和下单。该流程适用于在已有panel基础上添加额外探针(最多100个基因),或者人或小鼠物种标准基因的单独定制(最多480个基因)。
2)高级定制流程(Advanced workflow):该流程需要先联系10x Genomics的销售,确定好订单,然后通过Xenium Panel Designer软件提交需求,并和10x Genomics生信技术团队一起完成探针设计。该流程适用于非人和小鼠的物种(含多物种来源样本)以及特殊需求设计(如SNVs、等位基因、基因融合、病毒序列、细菌序列、荧光蛋白、转基因、CRISPR guides、CDR3克隆型、蛋白标签、barcodes等)。
下面我们将以标准流程为例,为您展示如何顺利完成一个Xenium探针panel的定制。
Xenium探针设计需要以注释好细胞类型的单细胞表达数据作为参考,来预测可能引起光学拥挤(某个荧光信号过高时,会减弱其他相对中等或偏弱的荧光信号的检出灵敏度)的高表达基因。对于单细胞测序数据的选择,Xenium Panel Designer软件支持以下三种数据来源:
注释好的单细胞测序数据
1、客户提供与待测样本相同或相近来源的单细胞测序数据,可接受HDF5、MEX或CLOUPE三种数据格式。
如果提供的数据格式为CLOUPE文件,应提供Cell Ranger生成的单个未压缩CLOUPE文件。Xenium panel设计工具使用基于图形的聚类结果进行细胞注释,因此,提供此类输入格式不需要额外的注释文件。
如果提供的数据是HDF5或MEX格式,要求提供一个或多个过滤了barcode的非归一化的全转录组矩阵,每个矩阵都要有细胞类型注释。矩阵和注释文件应打包成.zip, .tar, 或tar.gz文件(一个矩阵和一个注释文件压缩成一个包),并且注释文件与矩阵文件中的 barcode后缀和前缀必须完全匹配。其中:
1)Feature-barcode矩阵可以是Cell Ranger HDF5或Matrix Exchange(MEX)格式。HDF5矩阵是一个文件,MEX格式是包含三个文件(matrix.mtx.gz, barcodes.tsv.gz, features.tsv.gz)的文件夹。
2)细胞类型注释文件可以是CSV或TSV格式。文件格式是一个两列的文件,每列要有列标题,并且第一列必须是barcode,示例如下:

3)注意事项
不要把矩阵归一化或进行基因过滤。归一化或基因过滤会影响对光强拥挤程度的评估。如果矩阵包含总基因统计数据的子集,会使每个基因的表述出现偏差。
参考数据文件打包后,文件大小应<500MB。文件过大会导致panel设计超时。如果文件超过500MB,建议在矩阵中减少样本个数。
关于矩阵文件,可以选择将多个样本作为一个聚合矩阵上传,也可以将它们作为单独的参考数据集上传。如果提供了多个矩阵,则将评估所有矩阵中的细胞类型信息。
如果关注稀有细胞类型,推荐每个样本提供一个矩阵文件,不要整合。如果提供多个矩阵,则会评估所有矩阵中的细胞类型信息。
2、对于客户没有自己的单细胞数据,10x Genomics官网也提供了多个公开可用的单细胞表达数据集(见表1)
表1:10x Genomics提供的预构建好的单细胞参考数据集

3、可以将客户自己的数据集与公开数据一起使用。
基因列表
1、基因格式
Xenium Panel Designer软件要求上传的基因列表为基因名称和/或Ensembl ID,后面跟着探针集的数量(可选),每行用逗号分隔。以EGFR基因为例,可以选择以下几种格式中的一种:
EGFR
ENSG00000146648
EGFR,5
ENSG00000146648,5
EGFR, ENSG00000146648,7
ENSG00000146648, EGFR,7
2、基因格式的注意事项
基因符号和特征ID必须与人或小鼠2020-A版本的参考GTF文件中的特征完全匹配。
Ensembl ID不能包含“版本小数”(例如,“ENSG00000010404”是可以的,而“ENSG0000010404.3”则不可以)。
Ensembl ID必须是基因ID(ENSG、ENSMUSG),而不是转录本ID。
基因格式不符合要求,可能导致后续实验数据中基因比对失败。
3、关于基因列表的补充说明
列表中基因的排序:按照从最重要到不重要进行排序,有助于优化考虑在最终设计中保留的基因。
对于添加定制探针,最多可添加100个基因,添加的基因应不包含在已有panel中。
无论哪种panel类型,建议提供10-20个额外的基因,备用于在设计过程中需要更换主组中的任何基因。
利用在线软件设计探针
1、打开软件页面:
登录10x Genomics官网(需要提前注册好账号),然后打开Xenium Panel Designer在线设计软件(https://cloud.10xgenomics.com/xenium-panel-designer)。
2、提交数据:
按照下图所示workflow依次填写样本信息、选择待添加探针的已有Panel、上传单细胞数据、提交基因列表。
需注意,上传数据过程中,当输入文件存在以下问题时,会报错:
矩阵、基因列表和2020-A参考之间的基因ID和/或基因符号不匹配。
基因名称中包含空格/空白或拼写错误。
文件缺少列标题或标题中有特殊字符或拼写错误的名称。
矩阵和注释CSV文件的barcode不完全相同。当注释文件中的barcode在整合后有额外的样本后缀,但矩阵本身没有时,这种情况经常出现。
3、根据summary建议进行调整:
在正式提交(Finish)前,软件会先生成一个panel summary,对探针设计的各项细节给出评估。一般情况下,开始返回的panel summary中可能有多项风险提示,比如某些基因表达过高可能导致光学拥挤、部分细胞类型无法有效区分、部分基因无法找到足够的探针组等,此时,需要根据建议对基因列表进行调整后再重新尝试,有时需要反复多次调整后才能得到满意的panel summary(无风险)。
需注意,以下这些输入文件问题,不会影响panel设计流程,但会导致不好的结果:
归一化统计数据不会让设计软件报错,但会让结果出现偏差。
过滤掉许多基因的矩阵文件不会让设计软件报错,但结果会出现偏差,从而产生次优设计。
基因列表中存在基因缺失的矩阵文件。
基因表达数据不匹配。
注释CSV文件前两列不是“barcode,annotation”。如果层次注释存在于其他列中,则会被忽略。
4、正式提交:当完成设计调整,确认最终基因列表后,点击finish完成最终版本的提交,并会在稍后得到一个Design ID。
以上就是10x Xenium定制探针的标准流程,将Design ID提供给10x 产品经销商,就可以走后续的商务流程了。如此简洁丝滑的探针设计流程,你get了吗?






