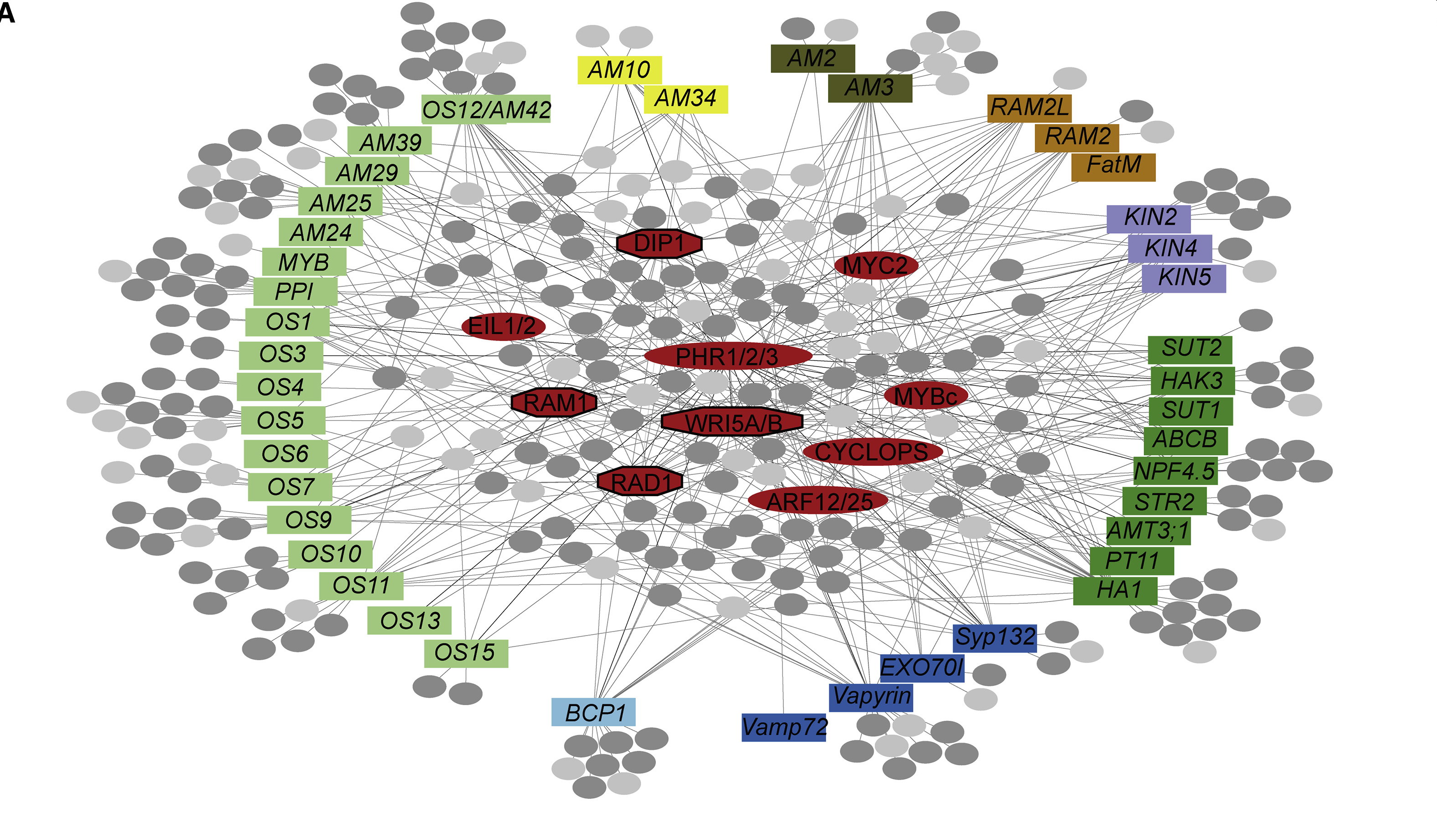
2021 年 10 月 12 日,中国科学院分子植物科学卓越创新中心王二涛研究员为通讯作者,在 Cell 上发表了题为“A phosphate starvation response-centered network regulates mycorrhizal symbiosis”的封面论文。文章首次报道了水稻-丛枝菌根共生转录调控网络,发现植物直接营养吸收途径与通过丛枝菌根共生的间接营养吸收途径均受到磷信号网络的统一调控,回答了菌根共生领域“自我调节”这一困扰领域的重要科学问题。
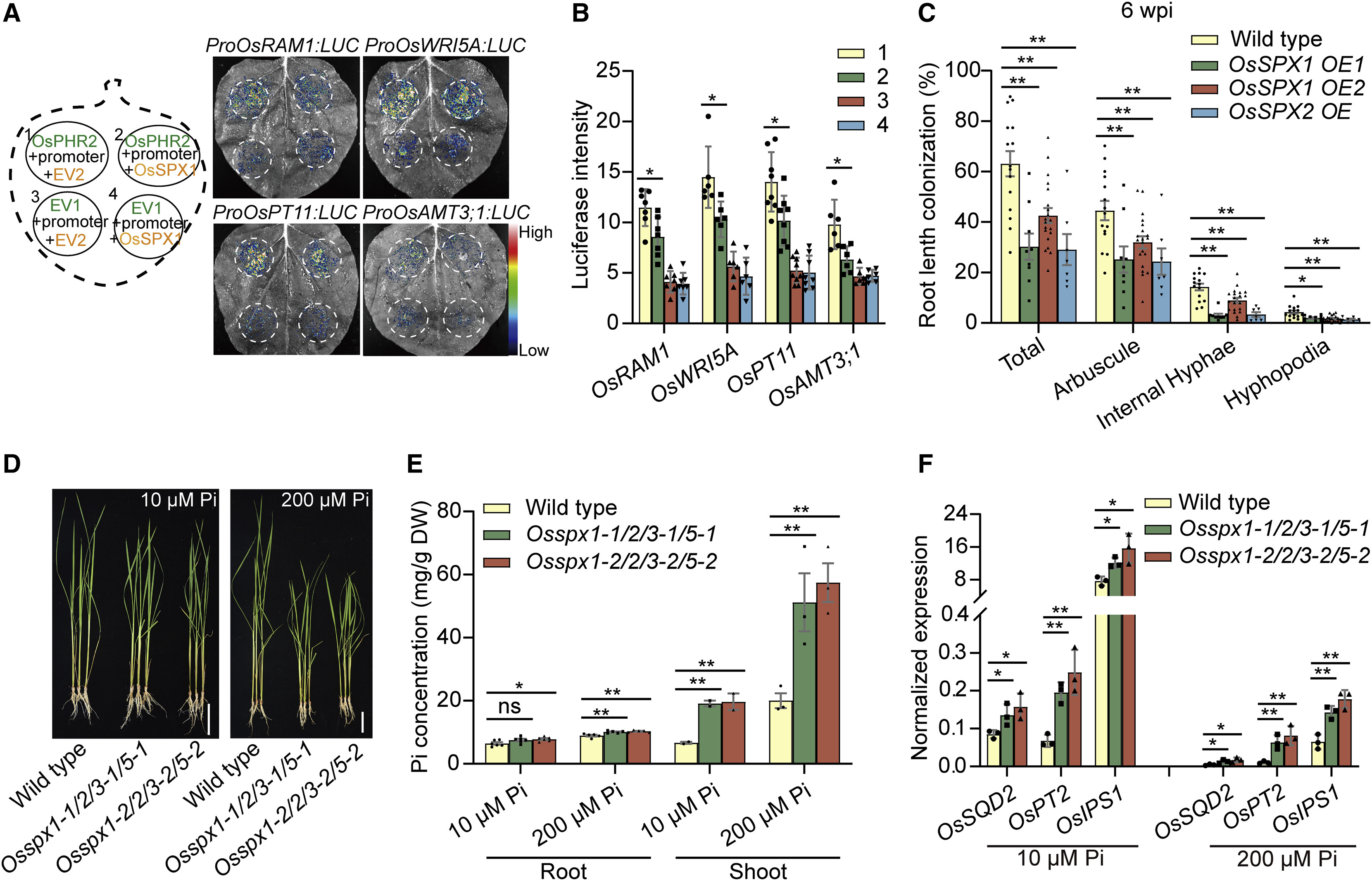
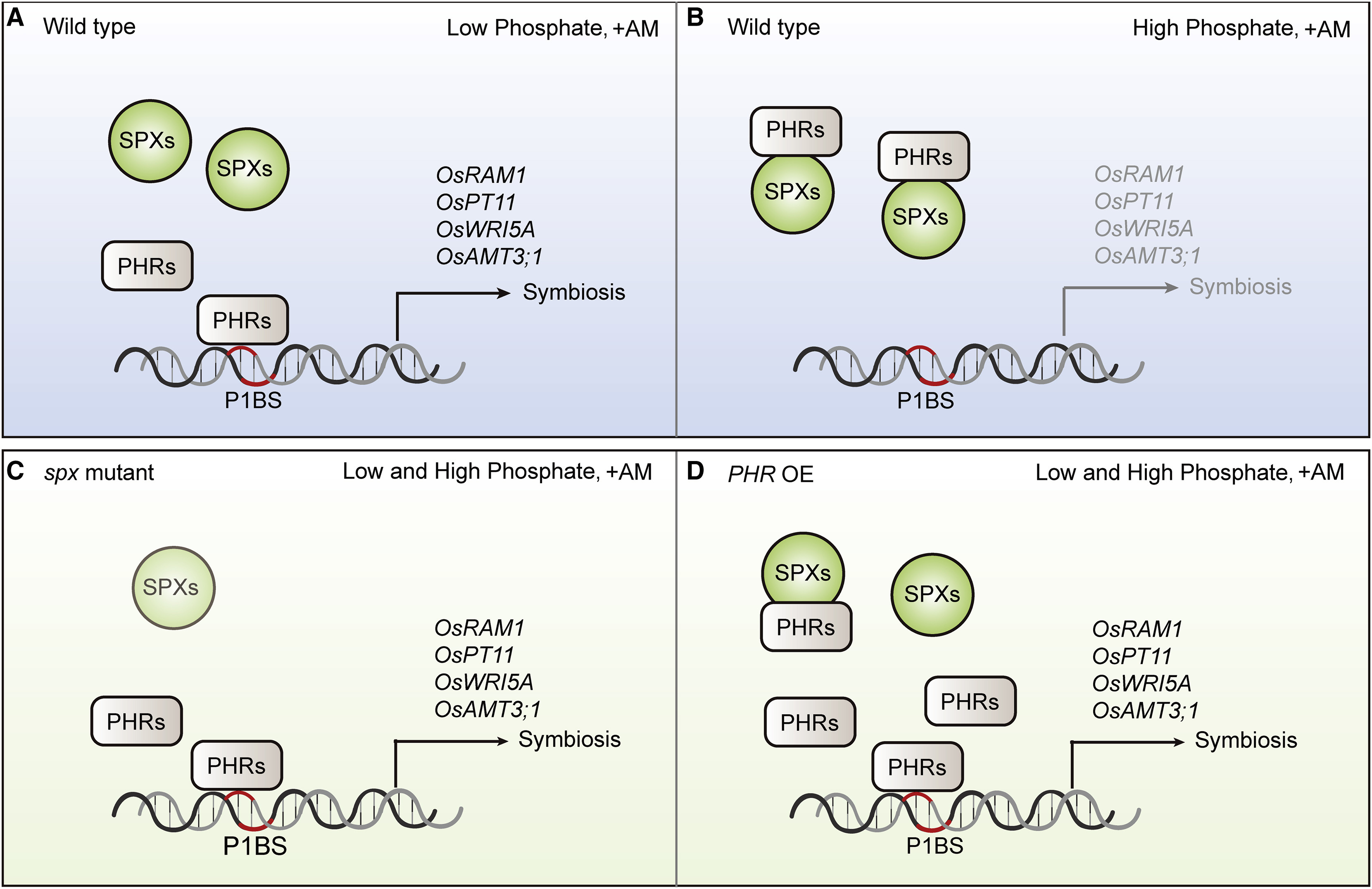
分子植物科学卓越创新中心博士后石进彩为第一作者,欧易生物肖云平也参与了此项研究。文章中水稻转录因子文库筛选服务由欧易生物提供。 研究背景 磷是植物生长发育所必需的养分之一,但土壤中的磷元素大多数以植物不可吸收的形式存在。为了提高对磷等营养的获取,陆生植物主要有两种途径获取营养:1)通过根系直接从土壤中吸收营养;2)与其菌根真菌共生,间接获取营养。 植物-丛枝菌根共生的建立,与植物由水生向陆生进化发生在同一时期,是植物适应陆地环境关键事件之一。丛枝菌根共生是自然界最普遍的一种共生,是植物从环境中高效获取营养的重要途径。 植物根据自身的磷营养状态调控其与丛枝菌根真菌之间的共生,研究人员称为菌根共生的“自我调节”(”self-regulation” nature of mycorrhizal symbiosis),但其调节机制未知。 研究内容 本研究利用水稻转录因子文库筛选,绘制了水稻转录因子和菌根共生相关基因之间的调控网络。该网络受到保守的磷感应途径统一控制,以磷酸盐饥饿响应因子(PHR)为中心。PHRs 是菌根共生所必需的,通过 P1BS 顺式作用元件调控菌根共生相关基因表达。高磷条件下,SPX蛋白抑制 OsPHR2 介导的对菌根共生相关基因表达的调控,进而抑制菌根共生。过表达 OsPHR2 可以部分拮抗磷介导的对菌根共生的抑制作用。本研究发现,植物直接营养吸收和通过菌根共生的间接营养吸收,都受到相同的磷信号网络统一调控。本研究将有助于高效用磷植株的培育。 研究结果 1. 水稻-丛枝菌根共生转录调控网络 为了系统地揭示调控丛枝菌根共生的转录因子,研究者以 51 个水稻基因的启动子为诱饵,利用包含 1570 个水稻全长转录因子的文库进行了酵母单杂交筛选。这 51 个基因主要是已报道的参与或调节菌根共生的基因。 文库筛选发现了一个由 266 个转录因子和 47 个启动子高度连接的网络,称之为“丛枝菌根共生酵母单杂交网络(YAM)”。平均每个启动子可以和4 个转录因子互作。266 个转录因子分属至少11 个转录因子家族。72%(192 个)的 YAM 转录因子在根中存在表达(FPKM > 1)。与非定殖根相比,19 个 YAM 转录因子在丛枝菌根真菌定殖的根中表达上调。 图片说明:水稻-丛枝菌根共生调控网络 2. YAM 节点在菌根共生中的作用 接下来利用RNAi植株 和水稻突变系验证上述 YAM 调控网络。 OsEIL1/MHZ6 和 OsEIL2 是乙烯信号转导所必需的,对应的突变体对乙烯不敏感。结果显示,与野生型相比,OsEIL2 RNAi 植株中丛枝菌根真菌的定殖率更高(图 C)。在 YAM 中,OsEIL2 可以调控菌根共生特异性 H+-ATPase OsHA1 的表达,因此推测乙烯信号可能会抑制菌根共生。 OsARF12 和 OsARF25 是磷酸盐稳态负调控因子,是水稻根系发育所必需的。与野生型相比,Osarf12/25 双突变体菌根共生率显著降低(图 D)。在 YAM 中,OsARF12/25 可以与 OsHA1 等基因启动子结合,但在 Osarf12/25 双突变体中 OsHA1 高表达,其机制有待进一步研究。 同样,敲除 YAM 中的 OsRAM1、OsWRI5a/b 也减少了菌根共生(图 E-F)。MYB 型转录因子 OsMYBc 可以直接靶向钾转运基因 OsHKT1 调控耐盐性,与野生型相比,OSmybc 突变体菌根共生水平也下降(图 G),表明耐盐性和菌根共生可能受到共同调控。 图片说明:调控网络中转录因子的功能验证 3. PHR 为中心的调控网络控制丛枝菌根共生 酵母单杂交结果显示,磷酸盐饥饿响应因子 OsPHR1/2/3 可以激活 51 个目标启动子中的 25 个,包括 OsRAM1、OsRAD1、OsWRI5A 等。OsRAM1、OsRAD1、OsWRI5A 自身又可以调控许多菌根共生相关基因的表达(图 A)。 对丛枝菌根共生响应基因启动子序列预测分析显示,有 78 个转录因子被富集,87% 的菌根共生响应基因受到 OsPHR1/2/3 的调控,其中 42% 受到 OsPHR1/2/3 的直接调控。因此推测 OsPHR1/2/3 可能是菌根共生调控网络中的一个关键调控枢纽(图 A-B)。 EMSA 和荧光素酶报告实验证实,OsPHR12可以直接结合并激活 OsRAM1、OsWRI5A、OsPT11、OsAMT3 启动子序列(图 C-F),并且这种结合依赖于启动子中的 P1BS 元件(图 G)。 OsPHR1/2/3 在菌根共生的根中表达量变化不显著,但 OsPHR2 在丛枝菌根真菌定殖的细胞中表现出高表达。与对照相比,Osphr2-1 突变体根中菌根真菌定殖减少(图 A-D),重建 OsPHR2 表达后突变体中菌根真菌定殖率与对照相当。转录组测序显示,OsPHR2 功能缺失改变了 63% 菌根共生相关基因的表达。反之,过表达 OsPHR1 和 OsPHR2 可以引起菌根共生的显著增加(图 E-G)。 以上结果表明,以 PHR 为中心的调控网络通过 PIBS 元件依赖的菌根特异性基因表达控制菌根共生。 图片说明:OsPHR2 是丛枝菌根共生所必需的 4. 磷酸盐通过以 PHR 为中心的网络调控菌根共生 植物的磷营养状况与菌根共生程度呈负相关。经一定浓度的 Pi 处理,与对照相比,OsPHR2 过表达植株丛枝菌根共生下降程度不明显(上图 E-G)。表明过表达 OsPHR2 可以对磷酸盐介导的菌根共生抑制作用产生部分拮抗。 SPX 蛋白参与磷酸盐稳态维持,在高磷条件下可以与 OsPHR2 结合以抑制 OsPHR2 对 P1BS 元件的结合。荧光素酶报告实验证实,OsSPX1 可以特异性抑制 OsPHR2 介导的 4 个菌根共生相关基因启动子的激活(图 A-B)。OsSPX1、OsSPX2 过表达可以显著抑制丛枝菌根共生(图 C)。与对照相比,osspx1/2/3/5 四突变体中丛枝菌根共生率显著上升。 以上结果表明,SPX 蛋白通过 PHR 为中心的调控网络负向调控菌根共生。 图片说明:SPX 蛋白通过 OsPHR2 负向调控丛枝菌根共生 研究结论 本研究首次报道了水稻-丛枝菌根共生的“自我调控”网络: OsPHRs 通过与 P1BS 元件结合直接调控菌根共生相关基因 OsRAM1、OsPT11、OsWRI5A、OsAMT3;1 的表达。SPX 蛋白 (磷酸盐传感器) 在高磷酸盐条件下与 OsPHR2 结合,抑制其与菌根共生相关基因启动子 P1BS 元件结合,从而降低了这些基因的表达,进而抑制菌根共生。PHR 过量表达植株和 SPX 缺失突变体的菌根共生对高磷处理不敏感,表明高磷通过 PHR-SPX 模块抑制菌根共生。 原文链接:https://doi.org/10.1016/j.cell.2021.09.030