2024年7月,中国海洋大学王师课题组在Communications Biology(IF=5.2)发表了题为“Tracking the hologenome dynamics in aquatic invertebrates by the holo-2bRAD approach”的研究论文,该研究建立了可高效实现对海洋无脊椎动物及其共存微生物即共生体基因组(hologenome)进行动态跟踪的holo-2bRAD,并在扇贝和对虾中进行了系统的方法学验证,同时利用该技术对双壳贝类“幼虫沉苗”现象进行了宿主-微生物联合解析。

发表期刊:Communications Biology
影响因子:5.2
DOI:10.1038/s42003-024-06509-7
涉及的欧易生物服务产品:2bRAD-M简化微生物组
研究背景
“共生体基因组”的概念是一种日益流行的思考海洋生物微生物和宿主的方式。然而,跟踪共生体基因组的动力学变化是具有挑战性的,这是由于跟踪动态变化需要大量的材料,以及跟踪本身通常会导致研究对象的损伤或死亡。本研究展示了一种简单和高效的holo-2bRAD方法,从一个holo-2bRAD文库来跟踪海洋无脊椎动物(即扇贝和虾)的共生体基因组动态。
研究内容
本研究提供了一种用于共生体基因组分析的方法,即holo-2bRAD。该方法可以概括为两部分:实验部分和分析部分(图1)。实验流程从非侵入性取样开始,选择合适的取样方法(如,用棉签擦拭鳃或用移液管收集粪便)获取材料,然后使用相应的DNA提取方法提取混合DNA。最后,按照文库构建方法制备一个holo- 2bRAD文库,然后进行下一代测序。
在分析流程中,首先需要建立由微生物和宿主的特异性2bRAD标签组成的共生体基因组数据库(“holo-DB”),然后将高质量测序reads与holo-DB进行比对,以确定用于宿主基因分型和微生物分析的可靠reads。最后,利用RADtyping对宿主进行基因分型和利用2bRAD-M简化微生物组进行微生物组成和相对定量分析。
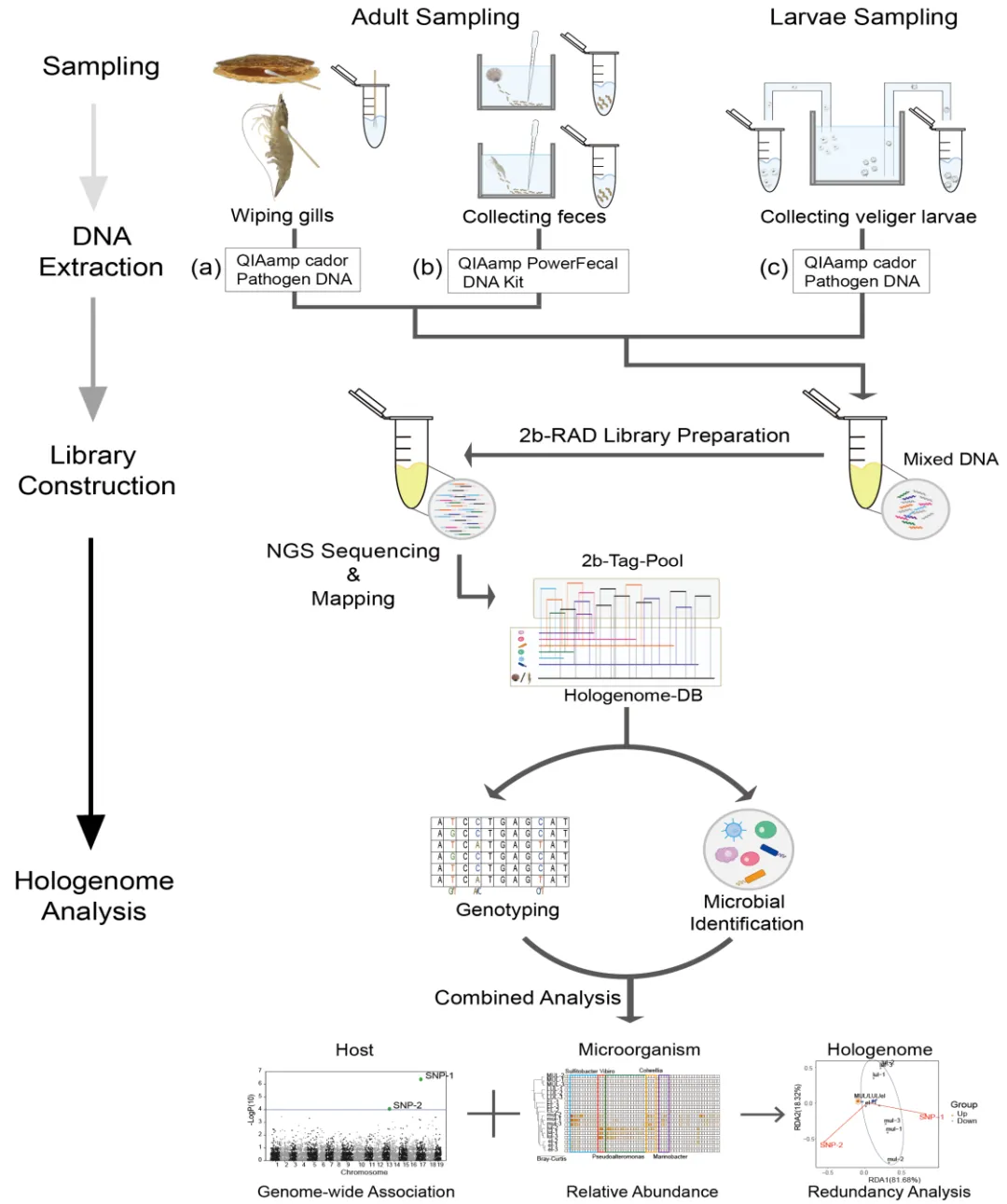
图1 扇贝和虾的holo-2bRAD共生体基因组分析流程总览
研究结果
1. 无创取样-取样材料的选择
采用革兰氏染色法评估全身多个组织,包括鳃、粪便、虾脚、肌肉、性腺、外套膜等,结果发现,鳃和粪便中细菌含量最高,是共生体基因组分析无创取样的最佳材料。
在确定了要取样的组织或材料之后,如何进行取样呢?在自然排泄后可以直接收集粪便,而如何快速、无破坏性地获得效率充足的鳃材料仍然是一个挑战。在本研究中,我们使用了一种无创擦拭方法,并评估了不同的擦拭材料,即滤纸和棉签,以评估选择哪种取样材料。棉签获得的DNA总量(1.45-2.97 µg)和质量优于滤纸(0.31–0.50 µg),选择棉签作为取样材料。
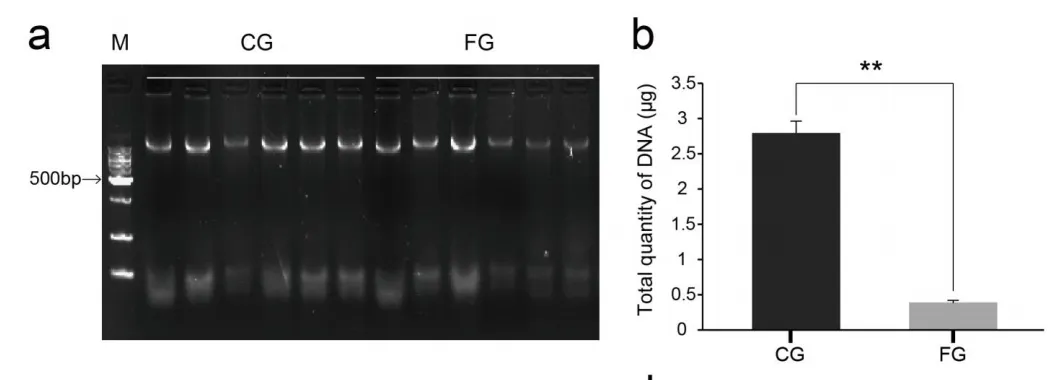
图2 用棉签(CG)、滤纸(FG)擦拭制备的DNA的琼脂糖凝胶电泳分析和DNA总量
2. 共生体DNA的提取
把V4-V5区域(400bp)和扇贝、虾的基因组进行比对,发现在此区域扇贝和虾也能比对上,扇贝~600bp,虾~590bp。扇贝共生体DNA的电泳分析显示有两条条带(400和600 bp),表明宿主和微生物的DNA同时存在,共生体DNA提取成功。
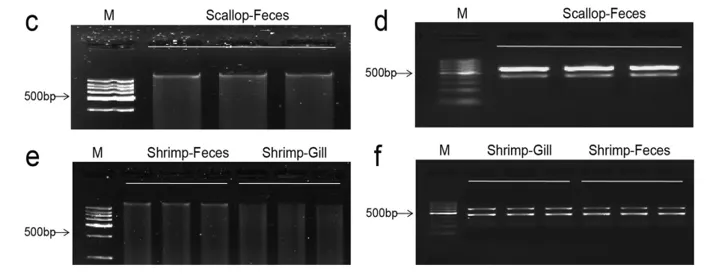
图3 共生体基因组DNA的琼脂糖凝胶电泳分析
3. 共生体基因组数据库(holo-DB)的构建
从NCBI RefSeq下载173165个微生物基因组,包括25260个细菌、614个古菌和289个真菌,用BsaXI对所有基因组进行酶切,保留在每个基因组只出现一次的微生物类群特异性标签,即holo-2bRAD标签。对宿主基因组(如扇贝和虾)进行电子酶切,去冗余后分别获得169582(46.2%)和653495(84.5%)个unique标签。最后,将微生物unique标签和宿主unique标签去除冗余,构建holo-DB。
在构建holo-DB的过程中,发现扇贝/虾和微生物之间共享的标签很少。比如,在扇贝的holo-DB中,只去除111个标签,占所有非冗余标签的0.07%;去除约8218个微生物基因组,占所有微生物基因组的4.75%。去除的微生物基因组在属到门水平上几乎没有影响(如0.01%至0.2%),只有影响3.71%的种水平。
4. 扇贝和虾的共生体基因组分析性能
首先,从基因组覆盖率、召回率、基因分型一致性和基因分型准确性四个方面评估了扇贝共生体基因组对宿主的性能。发现所有样本的覆盖范围一致(图4a);在技术重复中,每个标签的测序深度高度一致,皮尔逊相关系数 r>0.94(P<0.05)(图4b);在鳃和粪便样本中,重复间的基因分型一致性高,分别为99.86%和99.90%;基因分型准确性也较高(99.88%和99.87%);表明holo-2bRAD可以提供令人满意的基因分型。然后,评估了holo-2bRAD分析扇贝微生物群落的能力。在鳃和粪便样本中,两个技术重复之间检测到的物种数量具有很高的相似性(~95%)(图4c),相对丰度没有显著差异(图4e),高重现性表明共生体基因组分析的性能稳定。
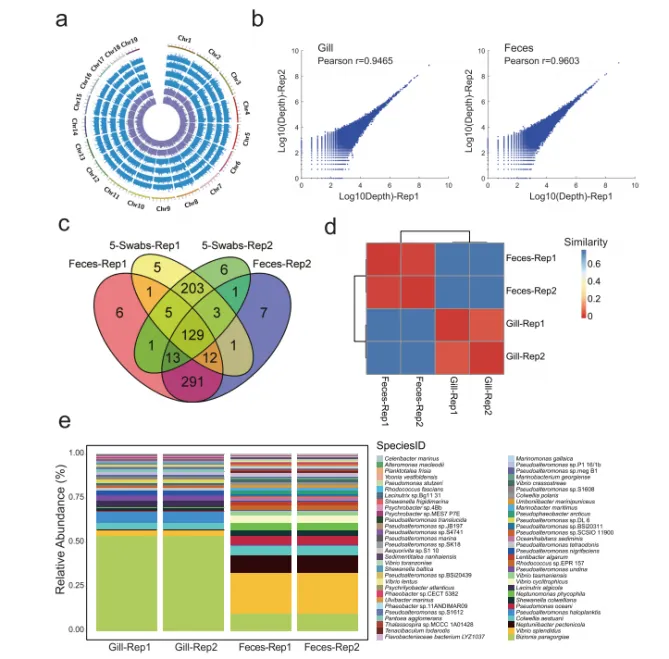
图4 扇贝共生体基因组分析性能
虾共生体基因组分析也包括宿主和微生物两部分。对宿主来说,把鳃和粪便共6个样本的所有SNPs构建系统发育树(图5a),同一个体的不同组织/材料聚集在一起,样本3为一个独立于其他个体的主要分支。虾中鉴定的微生物种类的层次聚类显示,相同类型的材料聚类在一起,而在同一组织/材料中,样本1和样本2更相似(图5b)。这些结果表明,尽管个体的年龄和生长环境相同,但个体间的微生物组成存在差异。宿主基因型相似的个体往往具有更相似的微生物组成,表明宿主遗传变异可能与之微生物群的分布相关。

图5 虾共生体基因组分析性能
5.不同存活力的扇贝幼虫的共生体基因组分析
从扇贝幼虫大量死亡的孵化场(MM)采集三个发育期的扇贝幼虫,它们的DNA具有低生物量或降解的特点,但我们成功构建了holo-2bRAD文库。通过比对到holo-DB,发现上层幼虫的细菌群落与下层幼虫的细菌群落差异显著;三个发育阶段的上层幼虫聚类在一起,下层幼虫根据不同的发育阶段明显分为三个聚类(图6a、b);来自于没有发生大量幼虫死亡的孵化场(NMM)的样品聚集在一起,没有发生分离(图6c、d)。另外,分析了在不同阶段丰度最高的种。在下层,top3的种都属于硫杆菌属,科尔韦氏菌属、海杆菌属在mul高,这三个属随着发育丰度都降低;相比下层,上层种的丰度显著降低,三个阶段间无显著差异。
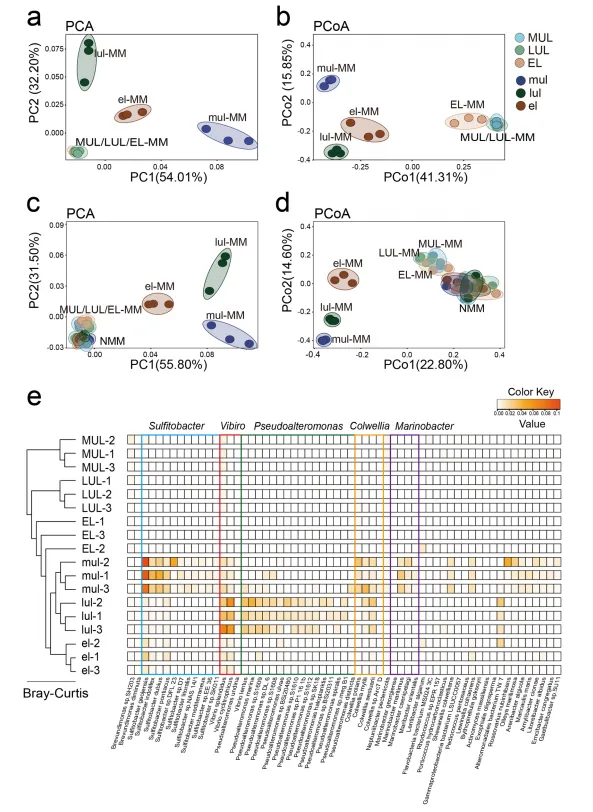
图6 扇贝体内微生物组成随幼虫生存能力的动态变化。大写代表上层的幼虫,小写代表底层的幼虫。MM是发生幼虫大量死亡的孵化场,NMM是未发生大量死亡的孵化场。
通过对扇贝幼虫进行基因分型,上层幼虫获得48820个SNPs,下层幼虫获得48174个SNPs,其中上下幼虫共有SNPs 43234个。通过GWAS分析,以确定与幼虫生存能力相关的潜在SNP。结果表明,有两个SNPs与幼虫的生存能力有很强的相关性。其中一个位于编码抗肌萎缩蛋白(Chr17)的基因的内含子区域,据报道该蛋白与扇贝肌肉的生长呈正相关。另一个位于甘露糖受体c型蛋白(Chr13)编码基因的基因间区域,该蛋白已被证明与扇贝的先天免疫呈负相关。通过冗余分析(RDA)证实了两个SNPs与相关微生物之间的相关性(SNP-1:R2 = 0.5069,P = 0.001;SNP-2:R2=0.4448,P=0.004)。
研究结论
本研究在前期2b-RAD技术(Nature Methods 2012;适用于宿主基因分型)和2bRAD-M®技术(Genome Biology 2022;适用于微生物定性和定量分析)的基础上,建立了可通过单文库实现动态共生体基因组(hologenome)分析的holo-2bRAD方法。该方法通过单次DNA提取和文库构建即可高效率实现宿主-微生物联合分析,而且对DNA投入量要求极低,无需破坏性取样,可动态追踪宿主-微生物的协同变化。
了解及咨询更多简化微生物组技术资料&前沿文献可添加小欧科服微信17317724501为您答疑解惑。






